The Present Perfect Tense
Поставьте глагол в форму Present Perfect, Past Perfect или Future Perfect.Our taxi… by 9 o’clock yesterday morning. Let’s...
Y-хромосома - специфический мужской фрагмент генетического кода, который избегает кроссинговера (перемешивания) и практически в неизменном виде передается от отца - к сыну, от сына - к внуку и т.д. Изучение Y-хромосомных гаплогрупп позволяет выяснить происхождение по прямой отцовской линии, вплоть до предковой популяции, у которой впервые распространился тот или иной Y-хромосомный маркер (мутация). Некоторые (как b_u_d_y_o_n ) идут и дальше, и пытаются связать принадлежность человека к той или иной гаплогруппе с определенным самосознанием и даже политической позицией. Это не слишком убедительно, поскольку в Y-хромосоме содержится лишь 2% генома, а остальная часть ваших генов может вообще не иметь отношения к предковой популяции, от которой вы унаследовали Y-хромосому. Гаплогруппа сама по себе - это не нация, не этнос и не раса.
Я из любопытства тоже сделал анализ на выявление Y-хромосомной гаплогруппы. Оказалось I2a2 . Что не удивляет, учитывая происхождение моих предков из юго-западных русских регионов, где эта гаплогруппа встречается довольно часто (ок. 15-20%). В целом, к четырем основным ветвям большой гаплогруппы I («варяжской» I1, «германской» I2b1, «славяно-балканской» I2a2 и «баскско-сардинской» I2a1) относится около 20% населения Европы. Это единственная гаплогруппа, которая зародилась непосредственно в Европе, ответвившись от предковой «кроманьонской» гаплогруппы IJ около 25 тыс. лет назад.
Распространение различных субкладов гаплогруппы I (Балкано-Украинское сгущение соответствует гаплогруппе I2a2)
Носители гаплогруппы I - это европейский аналог индейцев, потомки первого исконного населения Европы, относящегося к кроманьонской ветви сапиенсов. До них здесь жили только неандертальцы. Все остальные, включая арийцев (R1a, R1b) и финно-угров (N), «понаехали» в Европу уже потом, после окончания Ледникового периода. (Кстати, : картинка многокилометрового ледникового щита, доползающего чуть ли не до Киева, это фантастика)
Подробнее о палеоевропейцах написал b_u_d_y_o_n в своей книге «Реликтовая Европа» (pdf) . Несмотря на то что я отношусь скептически к попыткам выстраивать идентичность на базе гаплогрупп, некоторые умозаключения автора в моем случае оказались удивительно верными. Например, из его книги следует, что потомки палеоевропейцев должны иметь особо сильную склонность к регионализму и этнонационализму. Это естественно: если предки человека обитают здесь уже 1000 поколений, ему далеко не все равно, кто расхаживает по его исконной земле. «Аполлоническая» философия жизни палеоевропейцев мне тоже вполне свойственна. Собственно, о своей принадлежности к одной из ветвей палеоевропейской гаплогруппы я подозревал уже давно, поскольку всегда ощущал себя именно «аборигеном». Научно подтвержденное знание о том, что «отцы моих отцов» живут примерно в тех же краях уже 1000 поколений, еще со времен охотников за мамонтами, хорошо гармонирует с этим внутренним ощущением.
Карта современного доминирования основных гаплогрупп в Европе
Происхождение Y-хромосомной гаплогруппы I2a, ее эволюция и пути миграции популяций носителей этой гаплогруппы являются предметом моего специального изучения уже на протяжении 5 лет, начиная с того времени когда я увлекся активным изучением популяционной генетики человека.
Причина моего интереса к этой гаплогруппе довольно проста — генетический анализ моей Y-хромосомы (Y-STR + Y-снипы) показал, что с точки зрения филогенетической классификации она принадлежит к одной из ветвей этой гаплогруппы, и в силу этого банального биологического факта по своей прямой мужской линии я происхожу от древних представителей этой гаплогруппы. Менее важной причиной моего вовлечения в исследование этой гаплогруппы служит досадная малоизученность как I2, так и всей гаплогруппы I. Достаточно сказать, что последние специальные исследования по этой гаплогруппе были опубликованы в 2004,2006, 2007 годах — Rootsi, Siiri et al. (2004) , Rootsi, S. (2006), Peter Underhill et al (2007)). Остальные работы, особенно касающиеся Y-хромосомного разнообразия популяций на территории Балкан и бывшей Югославии, преимущественно повторяют выводы озвученные в вышеупомянутых трех работах, ничего не добавляя от себя (Marjanović, Damir; et al.2005, Peričić, Marijana et al. 2005; Rebała K., et al. (2007). Поэтому именно работы эстонской исследовательницы-попгенетика Роотси определили целый ряд основных положений-гипотез по вопросу происхождения гаплогруппы I, закрепившихся позднее в научных и научно-популярных (Википедия) источниках. К сожалению, значительная часть из этих гипотез оказалась ложными.
Ниже мы приведем содержащиеся в Википедии-статье о I2 некоторые положения общепринятые в официальной консервативной популяционной генетики, причем, судя по статье, некоторые из приведенных положений были позднее переосмыслены и развиты в окологенетических кругах любителей.
Наивысшей концентрации гаплогруппа I2 достигает у жителей Боснии , Сербии и Хорватии - ныне эти народы говорят на славянских языках, тем не менее концентрация «индоевропейской» гаплогруппы R1a у них минимальна, что позволяет видеть в них прямых потомков дославянского субстрата, каковыми на Балканах были иллирийцы и фракийцы , а также их предки - носители культуры Балканского неолита (в том числе и Трипольской культуры ). Последний тезис подтверждается тем фактом, что как балканский неолит является развитием ближневосточного, так и балканская гаплогруппа I родственна ближневосточной гаплогруппе J , хотя данные об их расхождении разнятся.Также широкое распространение данная гаплогруппа имела в центральной Европе эпохи Бронзового века (Культура полей погребальных урн : Пещера Лихтенштейн), что, однако, несколько пошатнуло традиционные представления о том, что в то время Европу населяли кельты - носители либо «кельтской» гаплогруппы R1b , либо «арийской» R1a . Родство балканской гаплогруппы I2 и скандинавской гаплогруппы I1 , при разорванности их популяций позволяет сделать вывод о том, что они были рассечены вторжением народов «кельтской» (а точнее иберийской) гаплогрупп.
Оставляя в стороне вопрос о связи между гаплогруппой и языком, гаплогруппой и археологической культурой (эти связи заслуживают отдельного исследования), мы увидим что главным аргументом Роотси в пользу балканской прародины I является значительное присутствие этой гаплогруппы в районе Динарских Альп/Балкан. Самое простое и дешевое с точки зрения парсимонии объяснение такой численности состоит в допущении преемственности части генофонда населения Балкан со времен последнего ледникового максимума в Европе (20 тыс. лет назад) т.е со времен балканского ледникового убежища. Как известно, географическое происхождение гаплогрупп Y-хромосомы можно установить по следующему эмпирическому критерию: в том месте или популяции, где возникла данная гаплогруппа, ее частота и STR-дисперсия (или возраст STR-изменчивости) максимальны по сравнению с другими популяциями (Sengupta et al., 2006). Эта простая, и казалось бы, логичная схема при более детальном рассмотрении на примере гаплогруппы I не выдерживает критики в свете новых данных. Стоит, однако, помнить что установка в статьях Роотси была задана в те времена, когда ученные-генетики имели только общее представление о структуре гаплогруппы I. Последующие исследования профильных ученных-популяционных генетиков вкупе с самоотверженным любительским изучением гаплогруппы I в коммерческих проектах FTDNA (Family Tree DNA — Haplogroup I Y-DNA Project, FTDNA I1 yDNA Haplogroup , FTDNA I2*New ISOGG I2b , FTDNA I2a Y-Haplogroup , FTDNA I-M223 Y-Haplogroup , FTDNA I2a2b-L38) и особо значимых усилий отдельных исследователей-любителей в так называемой ДНК-генеалогии (Kenneth Nordtvedt, Bernie Cullen, Steve Trangsrud, William Hartley, Yme Drost, Aaron J. Hill, Peter Hrechdakian, Hovann Simonian, Bob May, Vadim Verenich, Bill Morrow, Lawrence Mayka, Vladimir Semargl etc.) позволили лучше и глубже понять филогенетические отношения и структуры ветвей-субклад гаплогруппы, а также дать хронологические и географическую привязку по отдельным ветвям-субкладам гаплогруппы. Данное исследование ограничится лишь кратким образом современного состояния наших знаний о происхождении и ‘эволюции’ гаплогруппы I2a.
Происхождение и пути миграции гаплогруппы I2a.
В силу фрагментарности обзора мы не будем касаться не менее важных вопросов о том, как и когда гаплогруппа I появилась в Европе, а также вопросов о времени и месте разделения базальной ветви I на гаплогруппы I1 и I2. В настоящее время мы не располагаем достаточным количеством фактов в пользу одной из многочисленных и равновероятных версий сценария, и поэтому просто обойдем эти вопросы стороны в надежде на предстоящее изучение ДНК жителей Европы среднего палеолита.
Вместо этого мы начнем описание с представления филогенетического (‘родословного’) древа гаплогруппы I2a, оставляя в стороне остальные ветви этой группы. Вместо стандратных простых схем гаплогруппы I мы предлагаем каноническую версию древа в обработке Нордтведта (версия 2013 года). Основным ее преимуществом является наглядное изображение взаимного топологического расположение ветвей субкладов и внутрикладовых кластеров гаплотипов. Кроме того, приведенная под деревом шкала построена с учетом интеркладового времени дивергенции (расхождения) ветвей, вычисленных по методу кросс-дисперсии Кеном Нордтведтом. Наконец, графическое представление филогенетического древа дополняется конъюнктурной реконструкцией движения носителей гаплогруппы I в хронотопе доисторической Европы.
Хронология разделения гаплогруппы I2a выглядит следующим образом (наша версия):
Около 14 000 -15 000 лет тому назад из I2a
L460/PF3647/S2381
выделилась ветвь I2a2-L35/PF3862/S150, L37/PF6900/S153, L181, M436/P214/PF3856/S33, P216/PF3855/S30, P217/PF3854/S23, P218/S32
, которая практически сразу разделилась на I2a2a-L34/PF3857/S151, L36/S152, L59, L368, L622, M223, P219/PF3859/S24,
P220/S119, P221/PF3858/S120, P222/PF3861/U250/S118, P223/PF3860/S117, Z77
(ок.12 000 — 8 000 лет) и I2a2b L38/S154, L39/S155, L40/S156, L65.1/S159.1, L272.3
(ок.12 000 -8 000 лет).
Параллельно с этим из гаплогруппы I2a выделилась подветвь I2a1-P37.2 (15 000-10 000 лет до настоящего времени). Примечательно, что в ходе расширенного анализа был выделена отдельная гаплогруппа I2a1-F (определяющий не включенными в ISOGG древо снипами L1294 L1297 L1298). По вычисленным Нордтведтом интеркладовым возрастам, этот кластер выделился позднее выделения I2a1a, но раньше выделения I2a1b (т.е посередине между двумя ветвями). По мнению Нордтведта, последний общий предок I2a1 -F и I2a1a мог жить не позднее 20 000 лет до нашего времени. Характерно, что представители I2a1*-F живут преимущественно на территории современной западной Франции. Кроме того, в Западной Европей живут и представители I2a1c.
Из I2a1-P37.2 в период между 20 000 и 15 000 годами до насто ящ его времени выделились I2a1a L158/PF4073/S433, L159.1/S169.1, M26/PF4056((время жизни последнего общего предка — — 8 000- 6 000) и I2a1b-M423 L178/S328, M423 (время жизни последнего общего предка — не меньше 14 000 лет до настоящего времени), из I2a1b-M423 L178/S328 по интеркладовому счету около 13 500 лет выделились предковые линии I2a1b3 и I2a1b2 — (Dinaric+Disles) и Isles . Интересно, что время жизни последнего общего предка первых мы оцениваем примерно в 6600 лет до настоящего времени у Isles примерно 8000 лет.

Бо лее поздние внутрикладовые разделения (на у ровне 8 тыс.-6 тыс. лет до настоящего времени) неплохо коррелируют с реконструированным палеоклиматологами глобальным похолоданием ок. 6200 года до нашей эры, что привело к к отмеченному специалистами демографическому кризису неолита, когда количество народонаселения в Европе явно сократилось. На севере Европе в то же са мое время (примерно в 6500 -6200 году д.н.э) произошло событие, географически разделившее (филогенетически разделившиеся уже ранее) популяции I2a1b2-Isles и I2a1b3 , а также часть субклада I2a2-M284 – Isles от остальных субкладов I2a2. Речь идет об затоплении Доггерланда. Определенные с видетельства, в том числе контуры современного морского дна, показывают, что в эпоху последнего оледенения водораздел между Северным морем и Ла-Маншем проходил от Восточной Англии на юго-восток до Хук-ван-Холланда, а не через Па-де-Кале, и что реки Темза, Маас, Шельда и Рейн, соединяясь, текли по течению Ла-Манша в виде широкой реки, которая в конце концов впадала в Атлантический океан.[ Около 8000 г. до н. э. северное побережье массива суши, Догге рленд, представляло собой береговую линию с лагунами, болотами, приливными берегами и пляжами. Возможно, в эпоху мезолита эта территория была богатейшей в Европе с точки зрения охоты, добычи птицы и рыболовства.
Предполагается, что постепенный подъём уровня моря в результате таяния ледника (окончание последнего оледенения) привёл к затоплению Доггерленда Северным морем, что постепенно отрезало доисторическую Британию от европейского континента около 6500 г. до н. э. На месте будущей песчаной отмели, известной в настоящее время как Доггер-банка , согласно данной гипотезе, продолжал существовать остров по крайней мере до 5000 г. до н. э.
Согласно альтернативной, выдвинутой недавно гипотезе, большая часть данной земли была затоплена цунами около 8200 лет назад (6200 г. до н. э.), вызванным одним из крупнейших в истории оползней - дрейфом области подводного грунта от побережья Норвегии , известным как Стурегга . Согласно этой теории, цунами, вызванное оползнем, сыграло катастрофическую роль для мезолитического населения данной местности. В результате цунами, как представляется, Британия была окончательно отделена от континента, и, с культурной точки зрения, мезолит на ней пошёл своим путём. Одним из побочных результа тов оползня стало глобальное похолодание 6200 лет до н. э. ввиду притока холодных вод от растаявших ледников.
Кем были доисторические представители гаплогруппы I2a?
I. Где появились предки гаплогруппы I2a ?
Как видно из приведенной выше цитаты из Википедии, в среде некоторых окологенетических кругов укрепилось неверное на наш взгляд мнение о том, что I2a cвязана с кругом культур балканского неолита и в том числе, c культурами близкими к трипольцам. Такой вариант возможен, однако окончательный вариант должны дать анализы Y-хромосом древнее ДНК останков представителей этих культур. Но, если I2 и присутствовали у трипольцев, их вряд ли можно считать коренным элементом (ядро которого составляли выходцы с Ближнего Востока), скорее всего они представляли остатки местного мезолитического элемента, инкорпорированного в состав этой культуры. Если говорить об исконной культуре древних I2a2, то это скорее всего были свидерцы, и их эпигоны.
В 2011 году мне удалось немного подискутировать с администратором польского FTDNA проекта (Ларри Майкой) по вопросу о происхождении гаплогруппы I2a1b-M423 (и I2a в целом). До этого Майка относительно мало интересовался происхождением этой гаплогруппы (хотя, также как и я сам принадлежит к ней). Он предложил крайне интересную версию о месте происхождениия гаплогруппы I2a1. По его мнению, общий предок всех I2a1 (как I2a1*, I2a1a, I2a1b, I2a1c) c высокой степенью вероятности мог жить в районе Альпийских гор.
Он также согласился с предложенной мною маршрутом перехода клана I2a1 из Альпы в Карпаты. Правда, он не был согласен с моей датировкой. Я считаю, что предковая популяция I2a1 начала свое движение из Альп в сторону Карпат (из Западной Европы на север вслед за отступающим ледником), сразу после окончания ЛГМ (т.е. в мезолите где-то после 10 000 года до настоящего времени), Майка же считал что инициатором миграции могли быть кельты (то есть эта миграция произошла уже в бронзовом веке). Последний вариант хоть и интересный, но не объясняет времени и места разделения островного субклада I2a1b2 и динарского субклада I2a1b3, которая по молекулярной датировке не могла произойти позднее чем 10-8 тысяч лет тому назад.
С целью проверки версии Майки, я решил использовать алгоритм Мескита, который позволяет производить оценку наиболее вероятного (с точки зрения парсимонии) места «основания» субклада, исходя из нынешней географической дистрибуции (географического распространения) двух смежных субкладов (в данном случае мы определяем место появления I2a1b1 на основании сопоставления географических координат таксонов-гаплотипов и топологии дерева I2a1b, которое укоренялось аутгрупп-корнем I2a1a).
Несмотря на то, что в настоящее время наибольшая плотность субклада I2a1a приходится на Сардинию, по хорошо аргументированному и эмпирически подтвержденному мнению Нордтведта, самые старые по возрасту кластеры I2a1a приходятся на северный регион Пиренейского полуострова, Альпы и частично южную Францию
.
Поэтому в качестве координат аутгруппа-корня (гаплотипа I2a1a) были приняты координаты географического центра Пиренейских гор
C это целью я научилсь наносить в Меските филогенетические деревья прямиком на географическую проекцию Северной Европы, в 2D-3D проекции.
Для примера 2 карты, построенные мной в Меските-Картографер е на основании структуры филогенетического дерева из 687 гаплотипов I2a1b-M423, выявленной путем нахождения 20 независимых совпадений 20 лучших (парсимонических) деревьев в TNT. Красным цветом обозначено реконструированное место происхождения — в данном случае это регион Альп.

Таким образом полученная нами траектория движения носителей I2a1b примерно соответствует предложенному нами маршруту. Когда именно произошла миграция, сказать трудно так как между временем появления I2a1b и I2a1b3 прошло несколько тысяч лет. Важно другое, мы показали что движение началось не с территории Балкан, а скорее французских или швейцарских альп на северо-восток.
Подтверждается ли эта версия материальными свидетельствами — древней ДНК и пр.?
Буквально до самого недавнего времени, предложенная нами гипотеза оставалась лишь статистической интерполяцией нуждающиеся в дополнительных подтверждениях.
Пару недель была, наконец-то опубликована работа ‘профессиональных’ генетиков в которой показано как соотносятся большая часть из исследованных к настоящему времени ДНК древних образцов и ДНК современных популяций людей (Ancient human genomes suggest three ancestral populations for present-day Europeans). Мое предположение о исходной точке происхождения и начала миграций I2a1b, которое озвучивал на протяжении последних лет, получило в работе материальное подтверждение.
Во-первых, по однородительским линиям древний ДНК-генофонд охотников-собирателей шведского и люксембургского мезолита оказалось представителями мтДНК-гаплогруппы U и Y-хромосомной гаплогруппы I, так что опять мы имеем дело с отсутствием R1a/R1b у ранних европейцев мезолита.
Во-вторых, данные исследования показывают что юксембургский охотник-собиратель эпохи мезолита (чьи останки были найдены во время исследования мезолитической стоянки Хеффинген-Лошбур, Люксембург, примерно 8000 лет до настоящего времени) приндлежал к гаплогруппе I2a1b* M423 + L178+ . Согласно приведенным в таблице данным полиморфизмов Y-хромосомы, гаплогруппа это доисторического люксембургца является предковой по отношению как I2a1b2, так и I2a1b3
I2a1b L178 G A 15574052 12 +
I2a1b M423 G A 19096091 13 +
I2a1 P37.2 T C 14491684 7 +
I2a L460 A C 7879415 7 +
I2 M438 A G 16638804 14 +
I2 L68 C T 18700150 12 +
I P38 A C 14484379 2 +
I M170 A C 14847792 14 +
I M258 T C 15023364 5 +
I U179 G A 16354708 9 +
I L41 G A 19048602 3 +
В-третьих, данные исследования показывают также, что часть мезолитической популяции Мотала (Швеция, Эстгетерланд, примерно 8000 лет до настоящего времени) также принадлежала I2a1b*(xI2a1b1, I2a1b3)
Haplogroup I2 is the most common paternal lineage in former Yugoslavia, Romania, Bulgaria and Sardinia, and a major lineage in most Slavic countries. Its maximum frequencies are observed in Bosnia (55%, including 71% in Bosnian Croats), Sardinia (39.5%), Croatia (38%), Serbia (33%), Montenegro (31%), Romania (28%), Moldova (24%), Macedonia (24%), Slovenia (22%), Bulgaria (22%), Belarus (18.5%), Hungary (18%), Slovakia (17.5%), Ukraine (13.5%), and Albania (13.5%). It is found at a frequency of 5 to 10% in Germanic countries.
If you are new to genetic genealogy, please check our Introduction to phylogenetics to understand how to read a phylogenetic tree.
The phylogenetic tree of I2 evolved a lot over the last 10 years and top-level subclades were renamed more often than for any other haplogroup except R1b. To avoid all confusion it is recommended to double-check the defining mutations (SNP"s) when reading older studies or making references to a particular subclade. Several minor subclades were not included due to lack of space.
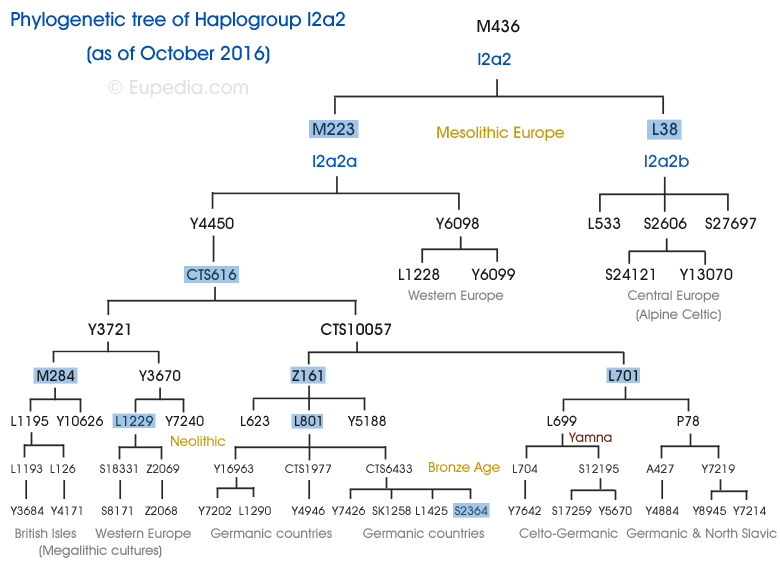
Click to enlarge

Haplogroup I is the oldest major haplogroup in Europe and in all probability the only one that originated there (apart from very minor haplogroups like C1a2 and deep subclades of other haplogroups). Haplogroup IJ would have arrived from the Middle East to Europe some 35,000 years ago, then developed into haplogroup I soon afterwards. It has now been confirmed by that the first Homo sapiens to colonize Europe during the Aurignacian period (45,000 to 28,000 years ago), belonged to haplogroups CT, C1a, C1b, F and I.
I2 (M438/P215/S31) is thought to have originated during the Late Paleolithic, around the time of the Last Glacial Maximum (LGM) , which lasted approximately from 26,500 to 19,000 years ago. I2 probably appeared in western Europe, although its exact region of origin cannot be determined since Paleolithic Europeans were nomadic hunter-gatherers. The oldest I2 sample recovered from archeological skeletons is a 13,500 year-old man from the Grotte du Bichon in Switzerland associated with the Azilian culture (see Jones et al. (2015)). His maternal lineage was U5b1h. As of October 2016, 15 Mesolithic European Y-DNA samples have been tested. Among them one belonged to haplogroup C1a2 (in Spain), one to F (in Germany), two to I* (in France), and six to I2 (Luxembourg and Sweden), including to I2a1* (P37.2), I2a1a1a (L672), I2a1b (M423) and I2c2 (PF3827). The maternal (mtDNA) lineages that they carried were U2e, U4, U5a1, U5a2 and U5b. The four samples from Russia belonged to Y-haplogroup J*, R1a1 (2x) and R1b1a. This shows that there was already a certain amount of diversity among Mesolithic European lineages, although many of these lineages (C1a2, F, I*, J*) are now exceedingly rare. Haplogroup I2a1 seems to have come out of the Neolithic period as the big winner for reasons that are not yet clear.
The analysis of Early Neolithic genomes from Anatolia by Mathieson et al. (2015) shows that I2c (L596) was already present in north-western Turkey 8,500 years ago, and had been assimilated by the Neolithic farmers on their way from the Fertile Crescent to Europe. Various conducted between 2014 and 2016 revealed that all Mesolithic European lineages became absorbed by the wave of Neolithic farmers. Yet I2a stood out as the most common among them, amounting to 16% of the 69 Neolithic Y-DNA samples tested. That places it as the second most common paternal lineage after G2a , the original lineage lineage of Anatolian farmers. I2a1 seems to have particularly prospered in the Starčevo–Kőrцs–Criş culture (6000-4500 BCE) in Southeast Europe, where it is still one of the main male lineages today. I2a1 also appeared in the Printed-Cardium Pottery culture (5000-1500 BCE) in the western Mediterranean. The modern Sardinians and Basques inherited the biggest share of their genome from those Neolithic farmers from the Printed-Cardium Pottery culture.
Although only one I2a2 has been found during the early or middle Neolithic samples so far (a I2a2a-M223 from Spain), many of them showed up during the Copper and Bronze Ages. Copper and Bronze Age samples consist of one I2a2, two I2a2a and one I2a2a1 (CTS616) in Spain, one I2a2a1b1b2 (S12195) in southern Russia (Yamna culture), one I2a2a1 (CTS9183) and one I2a2a1a2a (L229) in Hungary (Vatya culture), six I2a2b (L38) in Germany (Unetice and Urnfield cultures). I2a1 samples from the Copper Age include an I2a1a1 from northern Italy (Remedello culture), an I2a1 from Hungary (Vatya culture). I2c2 was also found in the Unetice culture in Germany.
Haplogroup I2a1 is by far the largest branch of I2 and the one most strongly linked to Neolithic cultures in south-east, south-west and north-west Europe.

I2a1a (M26 , L158, L159.1/S169.1) was known as I1b2 until 2005, I1b1b in 2006-7, and I2a1 from 2008 to 2010. It is found in all Western Europe, and reaches maximum frequencies among the Sardinians (37.5%) and the Basques (5%), two population isolates. M26 is geographically restricted to the British Isles, the Low Countries, France, western Germany, Switzerland, Sardinia, Sicily, the west coast of Italy, Iberia and the Mediterranean coast of the Maghreb. The only M26 negative for the L160 mutation are confined to Ireland.
I2a1a-M26 was probably one of the main paternal lineages of the Megalithic cultures of western Europe during the Neolithic and Chalcolithic periods. I2a1a1a (L672) was already found in Mesolithic Sweden, which implies that I2a1a had a very wide distribution from Iberia to Scandinavia during the Mesolithic period. Later, they would have adopted agriculture by intermixing with Near Eastern newcomers.
I2a1b (M423 , L178) was known as I1b until 2007, and I2a2 from 2008 to 2010. The main subclade, representing over 90% of all M423 lineages is L621 and its subclade L147.2 . The other subclades are L41.2 (very rare) and L161.1 (found mostly in Germany and the British Isles).
This branch is found overwhelmingly in Slavic countries. Its maximum frequencies are observed among the Dinaric Slavs (Slovenes, Croats, Bosniaks, Serbs, Montenegrins and Macedonians) as well as in Bulgaria, Romania, Moldavia, western Ukraine and Belarus. It is also common to a lower extent in Albania, Greece, Hungary, Slovakia, Poland, and south-western Russia. I2-L621 (L147.2+) is also known as as I2a-Din (for Dinaric).
The high concentration of I2a1b-L621 in north-east Romania, Moldova and central Ukraine reminds of the maximum spread of the Cucuteni-Trypillian culture (4800-3000 BCE). No Y-DNA sample from this culture has been tested to date, but as it evolved as an offshoot from the Starčevo–Kőrцs–Criş culture, it is likely that I2a was one of its main paternal lineages, and a founder effect could have increased considerably its frequency. The Cucuteni-Trypillian culture was the most advanced Neolithic culture in Europe before the Indo-European invasions in the Bronze Age and seems to have had intensive contacts with the Steppe culture before the expansion of Yamna to the Balkans and Central Europe (see histories of R1a and R1b). From 3500 BCE, at the onset of the Yamna period in the Pontic-Caspian Steppe, the Cucuteni-Trypillian people started expanding east into the steppe of what is now western Ukraine, leaving their towns (the largest in the world at the time), and adopting an increasingly nomadic lifestyle like their Yamna neighbours. It can easily be imagined that Cucuteni-Trypillian people became assimilated by the Yamna neighbours and that they spread as a minority lineage alongside haplogroups R1a and R1b as they advanced toward the Baltic with the Corded Ware expansion. Alternatively, I2-L621 lineages could have lived in relative isolation from the mainstream Proto-Indo-European society somewhere around Ukraine, Poland or Belarus, then as the centuries and millennia passed, would have blended with the predominantly R1a populations around them. The resulting amalgam would have become the ancestors of the Proto-Slavs.
Nowadays, I2a1 is five to ten times more common than G2a in Southeast Europe, while during the Neolithic period G2a was approximately four times more common. What can explain this complete reversal? At one point in history, I2a1 lineages seem to have benefited from being on the winning side. Apart from a minor boost from (hypothetically) joining Yamna"s westward expansion to Europe, the principal determining event that allowed I2a1b-L621 to become a major Eastern European lineage was probably the Slavic migrations from the 6th to the 9th century CE. Most modern Eastern Europeans belonging to I2a1b fit into the L147.2 (aka CTS10228, CTS2180 or Y3111) subclade, which is thought to have arisen 5,600 years ago (just before the Yamna period and the Trypillian expansion into the steppe), but has a TMRCA of only 2,300 years according to Yfull . The minority of I2a1b-L621 individuals negative for L147.2 are all found around eastern Poland, Belarus and western Ukraine, suggesting that this is where this lineage survived since the Chalcolithic. The I2a1b-L147.2 subclade seems to have expanded very fast from 1900 years ago, which is concordant with the timing of the Slavic ethnogenesis, considering that it takes a few centuries before one man can have enough male descendants to start having an impact at the scale of a population. This I2-L147.2 ancestor would have such an impact on the burgeoning Early Slavic population, still small 2,300 years ago, but booming.
After the Germanic tribes living in eastern Germany and Poland, like the Goths, the Vandals and the Burgundians, invaded the Roman Empire, the Slavs living further east filled the vacuum. Following the collapse of the Western Roman Empire in 476, the Slavs moved in the Dinaric Alps and the Balkans. By the 9th century, the Slavs occupied all modern Slavic-speaking territories, apart from the eastern Balkans under the control of the Turkic-speaking Bulgars.
Nowadays northern Slavic countries have between 9% (Poland, Czech republic) and 21% (Ukraine) of I2a-L621, while southern Slavs have between 20% (Bulgaria) and 50% (Bosnia). The higher percentage of I2a-Din in the south is probably just due to another founder effect due to the fact that the South Slavs originated in western Ukraine, where the ratio of I2a to R1a was higher. Virtually all Dinaric I2a falls under the L147.2 branch, and the majority to the S17250 ramification, who descend from a common patrilinear ancestor who lived only 1,800 years ago.
Commonly known in genetic genealogy circles as I2-M423-Isles , L161.1 is found at highest frequencies in western Ireland (5-10%) and the Scottish Highlands (1-5%), but is also found at low frequencies (> 1%) throughout Central and Western Europe, from Latvia, Lithuania and Belarus to the British Isles, and from Scandinavia to north-western Spain. It has also been found in Albania, northern Greece, Bulgaria and Romania.
The oldest known I2a1b-L161.1 individuals are the 8,000 year-old Loschbour man from Mesolithic Luxembourg, and a 7,800 year-old man from Motala in southern Sweden. I2a1b-L161.1 was probably scattered over most of Europe during the late Glacial and immediate postglacial periods, and in all likelihood integrated Neolithic society like all other Mesolithic lineages. I2a1b-L161.1 was very possibly one of the main Neolithic lineages in the British Isles during the Megalithic period, and would therefore have been among the men who built Stonehenge, Knowth, Newgrange and other great Megalithic sites. Its low frequency today and its confinement to the north-western and south-eastern fringes of Europe is indubitably the result of the numerous waves of Indo-European migrations over the last 5,000 years. The Proto-Celtic haplogroup R1b-L21 is now known to have arrived in Ireland around 2000 BCE (see Cassidy et al. 2015), only a few centuries after R1b first arrived in Central Europe. The transition was rapid, with R1b tribes overwhelming the Neolithic population of the British Isles in only a few centuries and pushing them further west and north, where L161.1 survives today.
These minor subclades are found at low frequencies mainly in north-Western Europe. Like I2a2 (see below) these lineages are probably the descendants of central and north-west European hunter-gatherers.
L880 is a very rare subclade identified in Germany, Luxembourg and France.
L1286 originated during the Late Paleolithic period. Nevertheless, 99% of modern carriers belong to the L233 subclade and share a common patrilinear ancestor less than 2,000 years ago. L233 is most common in the British Isles, but is occasionally encountered in the Netherlands, Scandinavia, Germany, France and Poland.
L1294 is also very rare and has only been found in Germany, France and Britain.
I2a2 (S33 /M436/P214, P216/S30, P217/S23, P218/S32, L35/S150, L37/S153, L181) was known as I1c until 2005 and I2b until 2010. It is associated with the pre-Celto-Germanic people of north-Western Europe, such as the megaliths builders (5000-1200 BCE). Its age has been estimated between 21,000 and 13,000 years old, which corresponds to the Epipaleolithic period.
I2a2 is found in most of Europe and seems to have had a continent-wide distribution before the arrival of Neolithic farmers. Several Mesolithic I2a2 samples have been identified to date, mostly by Mathieson et al. (2017) . This includes individuals from southern Germany (M223 from c. 7200 BCE), the Iron Gates between Serbia and Romania (Z161 from c. 6200 BCE), Latvia (CTS10057 from c. 5500 BCE) and southeast Ukraine (L699 from c. 5400 BCE and L701 from c. 5200 BCE).
Additionally, the subclade I2c1 (L1251) has recently been identified. It is found mostly in Germany and England and in their periphery (Ireland, Norway, France, Italy, Poland). This subclade is thought to be approximately 6000 years old, which places it in the Late Neolithic period. It may be associated with hunter-gatherers from Germany who adopted agriculture after coming into contact with Near-Eastern farmers from the Linear Pottery culture .
Andrew Johnson (1808-1875), the 17th President and 16th Vice President of the United States was identified as a member of haplogroup I2a2a (former I2b1) based on the results from the
111-маркерные гаплотипы были взяты как наиболее детальные, и оказалось, что среди них имеются 15 гаплотипов с первыми 12-маркерами точно такими же, как в представленном гаплотипе. Это гаплотипы с номерами 57-60, 110, 131, 132, 153-157, 660, 744, 745 по списку из 968 111-маркерных гаплотипов гаплогруппы I1. Далее было построено дерево из всех 968 111-маркерных гаплотипов, с помощью профессиональной компьютерной программы (PHYLIP), и наиболее родственные (по первым 12 маркерам) гаплотипы были выявлены. Полученное дерево приведено ниже, для иллюстративных целей. Оно показывает хорошую симметричность, что говорит о том, что все дерево происходит от одного общего предка. Когда он жил – будет также показано. Для сведения, базовый (предковый) гаплотип всего дерева следующий
13 22 14 10 13 14 11 14 11 12 11 28 – 15 8 9 8 11 23 16 20 28 12 14 15 16 – 10 10 19 21 14 14 16 20 35 37 12 10 – 11 8 15 15 8 11 10 8 9 9 12 23 25 15 10 12 12 16 8 13 25 20 13 13 11 12 11 11 12 11 – 32 12 8 17 12 24 27 19 11 12 12 13 11 9 11 11 10 12 12 31 11 13 21 16 11 10 24 15 19 11 24 17 13 15 25 12 22 18 12 14 18 9 12 11
Представленный гаплотип в первых 12 маркерах отличается от него всего на одну мутацию (пятая мутация по счету, выделено). Всего же все 968 гаплотипов показывают 14891 мутацию от первой 67-маркерной панели, и 24990 мутации на всех 111 маркерах. Это показывает, что общий предок жил 14891/968/0.12 = 128 → 147 условных поколений (по 25 лет), или 24990/968/0.198 = 130 → 149 условных поколений (0.12 и 0.198 – константы скоростей мутаций для 67- и 111-маркерных гаплотипов, стрелка – табличная поправка на возвратные мутации), или 3675±370 и 3725±375 лет до общего предка, соответственно. Видно, что 67- и 111-маркерные гаплотипы дают практически идентичные результаты, с разницей всего на 50 лет на фоне 3700 лет.

Оказалось, что указанные семь гаплотипов, содержащие такие же 12 маркеров/аллелей, находятся в трех разных ветвях дерева. Конечно, были бы в представленном гаплотипе 25, 37 маркеров или больше, определенно выявилась бы только одна ветвь, но что есть, то есть. В принципе, это нам помешает не сильно. Эти ветви – следующие:

Первая ветвь, в ней один гаплотип из перечисленных, под номером 58.

Вторая ветвь, включает гаплотипы 57, 744 и 745.

Третья ветвь, включает гаплотипы 59 и 60.

Последняя ветвь (из 19 гаплотипов) – сводная, она названа здесь четвертой, но включает полностью вторую ветвь (с гаплотипами 57, 744 и 745), плюс гаплотип 110. Базовые (предковые) 12-маркерные гаплотипы ветвей, соответственно:
13 22 14 10 13
14 11 14 11 12 11 28 — 1600±240 лет до общего предка
13 22 14 10 14 14 11 14 11 12 11 28 — 2960±410
13 22 14 10 14 14 11 14 11 12 11 28 — 1570±225
13 22 14 10 13
14 11 14 11 13
11 29 — 3150±350
(три мутации от представленного гаплотипа выделены). Представленный гаплотип в принципе может быть в любой из этих ветвей, его всего лишь 12 маркеров не позволяют исключить ни одну из четырех ветвей. Общие предки этих четырех ветвей жили, как указано справа от предковых гаплотипов.
Посмотрим, к каким субкладам относятся родственные представленному гаплотипы под приведенными выше номерами, и у которых полностью повторяются его первые 12 маркеров. В конце данного материала приводятся геномные данные по четырем из этих человек, в которых отмечены снипы, нижестоящие от M253. Скорее всего, эти же снипы будут у ID00020. В целом же снипы у большинства их этих гаплотипов: I1-M253 > DF29 > CTS6364 > M227 и в другой ветви I1-M253 > DF29 > Z58 > Z138. Снипы ID00020 должны относиться или к первой или ко второй цепочке. Все они будут определяться в московской Лаборатории ДНК-генеалогии.
Исторический путь к представленному гаплотипу примерно следующий. Гаплогруппа I образовалась около 43 тысяч лет назад (из сводной гаплогруппы IJ), около 28 тысяч лет она разошлась на гаплогруппы I1 и I2 (хотя «разошлась» здесь понятие условное, они не разошлись, это были независимые события), из первой (как и из второй) с течением времени образовалось множество субкладов. Гаплогруппа I была найдена в ископаемых скелетных останках с датировкой 7 тысяч лет в Швеции и Центральной Европе. В интервале 4600-4000 лет назад, в ходе заселения Европы эрбинами, носителями гаплогруппы R1b, гаплогруппа I1 из Центральной Европы практически полностью исчезла, но кто-то выжил, и как результат этого современные носители гаплогруппы I1 имеют относительно недавнего общего предка, который дал выживших потомков примерно 3700 лет назад, то есть в первой половине II тыс. до н.э. Поскольку общий предок в гаплогруппе I1 только один, дерево гаплотипов гаплогруппы столь симметричное. Такое встречается очень редко. ID00020 – потомок этого общего предка.
Приложение . Здесь приводятся детальные геномные данные по составу снипов, обнаруженных у носителей гаплотипов с ветви ID00020. У него должны быть практически такие, за исключением, возможно, терминальных снипов. Но, возможно, и они те же, как в нескольких приведенных случаях. Выделены снипы от М223 и ниже:
Kit No. 38184 (Matthew Hamilton Butcher, американский индеец, но получивший Y-ДНК от европейца, гаплотип 110 на ветви выше): DF29+ , M227+, CTS10058+, CTS10140+, CTS10338+, CTS10834+, CTS11036+, CTS11042+, CTS11441+, CTS11526+, CTS11552+, CTS11775+, CTS11783+, CTS11950+, CTS1393+, CTS2193+, CTS2375+, CTS2514+, CTS2524+, CTS2644+, CTS2738+, CTS3517+, CTS3654+, CTS3843+, CTS4088+, CTS4130+, CTS4295+, CTS4437+, CTS4848+, CTS4982+, CTS5167+, CTS5408+, CTS5513+, CTS565+, CTS5650+, CTS5705+, CTS571+, CTS5783+, CTS5884+, CTS5891+, CTS5908+, CTS5993+, CTS6140+, CTS6221+, CTS623+, CTS6265+, CTS6395+, CTS641+, CTS6629+, CTS674+, CTS6932+, CTS7267+, CTS7329+, CTS7831+, CTS7949+, CTS8333+, CTS8345+, CTS8394+, CTS8420+, CTS8716+, CTS88+, CTS8876+, CTS9240+, CTS9264+, CTS9288+, F1209+, F3692+, F719+, L118+, L121+, L123+, L124+, L125+, L132+, L15+, L16+, L187+, L350+, L403+, L41+, L468+, L470+, L498+, L509+, L574+, L575+, L578+, L740+, L748+, L75+, L750+, L751+, L755+, L756+, L758+, L759+, L772+, L80+, L81+, M168+, M170+, M213+, M227+, M235+, M253+ , M258+, M294+, M299+, M307+, M89+, M94+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P143+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P30+, P316+, PAGES00026+, PAGES00081+, PAGES00123+, PF2591+, PF2608+, PF2611+, PF2615+, PF2747+, PF2748+, PF2749+, PF2770+, PF3562+, PF3574+, PF3639+, PF3640+, PF3641+, PF3660+, PF3666+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3804+, PF3809+, PF3811+, PF3814+, PF3815+, PF3819+, PF3822+, PF3836+, PF3837+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, YSC0000281+, YSC0000301+, P19+, M42+, M450+, M72-, Z58-, Z63- , Z17694-, S2348-, S4442-, M3453-
Kit No. 38737 (Greathouse, Германия, гаплотип 744): CTS10058+, CTS10140+, CTS10338+, CTS10362+, CTS109+, CTS11036+, CTS11042+, CTS11358+, CTS11441+, CTS11526+, CTS11552+, CTS11575+, CTS11726+, CTS11775+, CTS11783+, CTS11950+, CTS125+, CTS12632+, CTS1393+, CTS1996+, CTS2193+, CTS2375+, CTS2514+, CTS2524+, CTS2536+, CTS2644+, CTS2738+, CTS3331+, CTS3431+, CTS3517+, CTS3536+, CTS3654+, CTS3662+, CTS3843+, CTS3868+, CTS3996+, CTS4088+, CTS4130+, CTS4295+, CTS4364+, CTS4368+, CTS4443+, CTS4740+, CTS4848+, CTS4982+, CTS5167+, CTS5318+, CTS5408+, CTS5457+, CTS5513+, CTS5532+, CTS565+, CTS5650+, CTS5705+, CTS571+, CTS5783+, CTS5891+, CTS5908+, CTS5993+, CTS6135+, CTS6140+, CTS6221+, CTS6265+, CTS6383+, CTS6395+, CTS641+, CTS6629+, CTS674+, CTS6800+, CTS6907+, CTS6932+, CTS7267+, CTS7329+, CTS7502+, CTS7831+, CTS7922+, CTS7933+, CTS7949+, CTS8243+, CTS8333+, CTS8345+, CTS8394+, CTS8420+, CTS8716+, CTS88+, CTS8876+, CTS8980+, CTS9240+, CTS9264+, CTS9288+, CTS9828+, F1046+, F1209+, F1302+, F1320+, F1329+, F1450+, F1460+, F1704+, F1714+, F1753+, F1767+, F2048+, F2075+, F2142+, F2155+, F2302+, F2345+, F2366+, F2402+, F2408+, F2587+, F2688+, F2710+, F2794+, F2837+, F2985+, F2993+, F3033+, F3111+, F3136+, F3335+, F3368+, F3402+, F3556+, F3692+, F4188+, F719+, F922+, L118+, L121+, L124+, L125+, L132+, L15+, L157+, L16+, L187+, L350+, L403+, L468+, L470+, L498+, L509+, L574+, L575+, L578+, L740+, L748+, L75+, L750+, L751+, L755+, L756+, L758+, L759+, L772+, L80+, M139+, M168+, M170+, M235+, M253+ Z138+ , Z139+, M170+, M258+, M307+, P19+, P30+, P38+, P109-, P259-, P37-, M161-, M72- , M26-, M21-, M223-, M227-
Kit No. 152077 (Jens Peter Nielsen, Дания, гаплотип 745): CTS10058+, CTS10140+, CTS10338+, CTS10362+, CTS109+, CTS11036+, CTS11042+, CTS11358+, CTS11441+, CTS11526+, CTS11552+, CTS11575+, CTS11726+, CTS11775+, CTS11783+, CTS11950+, CTS125+, CTS12632+, CTS1393+, CTS1996+, CTS2193+, CTS2375+, CTS2514+, CTS2524+, CTS2536+, CTS2644+, CTS2738+, CTS3331+, CTS3431+, CTS3517+, CTS3536+, CTS3654+, CTS3662+, CTS3843+, CTS3868+, CTS3996+, CTS4088+, CTS4130+, CTS4295+, CTS4364+, CTS4368+, CTS4443+, CTS4740+, CTS4848+, CTS4982+, CTS5167+, CTS5318+, CTS5408+, CTS5457+, CTS5513+, CTS5532+, CTS565+, CTS5650+, CTS5705+, CTS571+, CTS5783+, CTS5891+, CTS5908+, CTS5993+, CTS6135+, CTS6140+, CTS6221+, CTS6265+, CTS6383+, CTS6395+, CTS641+, CTS6629+, CTS674+, CTS6800+, CTS6907+, CTS6932+, CTS7267+, CTS7329+, CTS7502+, CTS7831+, CTS7922+, CTS7933+, CTS7949+, CTS8243+, CTS8333+, CTS8345+, CTS8394+, CTS8420+, CTS8716+, CTS88+, CTS8876+, CTS8980+, CTS9240+, CTS9264+, CTS9288+, CTS9828+, F1046+, F1209+, F1302+, F1320+, F1329+, F1450+, F1460+, F1704+, F1714+, F1753+, F1767+, F2048+, F2075+, F2142+, F2155+, F2302+, F2345+, F2366+, F2402+, F2408+, F2587+, F2688+, F2710+, F2794+, F2837+, F2985+, F2993+, F3033+, F3111+, F3136+, F3335+, F3368+, F3402+, F3556+, F3692+, F4188+, F719+, F922+, L118+, L121+, L124+, L125+, L132+, L15+, L157+, L16+, L187+, L350+, L403+, L468+, L470+, L498+, L509+, L574+, L575+, L578+, L740+, L748+, L75+, L750+, L751+, L755+, L756+, L758+, L759+, L772+, L80+, M139+, M168+, M170+, M235+, M253+ , M294+, M307+, M42+, M450+, M89+, M94+, P123+, P124+, P126+, P127+, P130+, P135+, P136+, P138+, P14+, P141+, P145+, P146+, P148+, P151+, P158+, P159+, P160+, P166+, P187+, P30+, P305+, PAGES00026+, PAGES00081+, PAGES00123+, PF1016+, PF1029+, PF1031+, PF1040+, PF1046+, PF1061+, PF1092+, PF1097+, PF110+, PF1203+, PF1269+, PF1276+, PF192+, PF210+, PF212+, PF223+, PF234+, PF258+, PF2591+, PF2593+, PF2599+, PF2608+, PF2611+, PF2615+, PF2624+, PF263+, PF2643+, PF272+, PF2745+, PF2747+, PF2748+, PF2749+, PF2770+, PF278+, PF292+, PF316+, PF325+, PF342+, PF3515+, PF3517+, PF3518+, PF3534+, PF3560+, PF3561+, PF3562+, PF3574+, PF3578+, PF3586+, PF3588+, PF3590+, PF3594+, PF3596+, PF3600+, PF3604+, PF3605+, PF3611+, PF3612+, PF3616+, PF3618+, PF3625+, PF3639+, PF3640+, PF3641+, PF3642+, PF3654+, PF3660+, PF3666+, PF3672+, PF3675+, PF3677+, PF3686+, PF3694+, PF3780+, PF3800+, PF3804+, PF3806+, PF3807+, PF3809+, PF3811+, PF3814+, PF3815+, PF3819+, PF3822+, PF3836+, PF3837+, PF500+, PF667+, PF719+, PF725+, PF779+, PF796+, PF803+, PF815+, PF821+, PF840+, PF844+, PF892+, PF937+, PF951+, PF954+, PF970+, V168+, V186+, V189+, V205+, V221+, V241+, V250+, V52+, V9+, YSC0000056+, YSC0000256+, YSC0000257+, YSC0000259+, YSC0000260+, YSC0000264+, YSC0000265+, YSC0000267+, YSC0000272+, YSC0000280+, YSC0000281+, YSC0000298+, YSC0000299+, YSC0000300+, YSC0000301+, Z138+, Z139+ , M170+, M258+, M307+, P19+, P30+, P38+, P109-, P259-, P37-, M161-, M72- , M26-, M21-, M223-, M227-
Kit N10060 (Robert Wade, Англия, гаплотип 57): M170+, M253+
, M258+, M307+, P19+, P30+, P38+, P109-, P259-, P37-, M161-, M72-, M26-, M21-, M223-, M227-
KLIN ID00004
Представлен 67-маркерный гаплотип, сообщено, что гаплогруппа I2a.
13 24 17 11 14 15 11 13 12 13 11 30 – 17 8 10 11 11 25 15 20 0 12 14 15 15 – 10 10 20 21 15 12 19 17 35 35 11 10 – 11 8 15 15 7 12 10 8 11 9 12 22 22 16 10 12 12 12 7 10 30 21 13 14 10 13 11 11 12 9
Эта гаплогруппа распространена по всей Восточной Европе, а именно в странах – Греция, Сербия, Босния-Герцеговина, Македония, Чехия, Словакия, Польша, Болгария, Белоруссия, Россия, Украина, Венгрия, а также немного в Германии, Румынии и Италии, и у подавляющего большинства тестированных был обнаружен снип I2a-CTS10228, он же L147.2 (см. диаграмму ISOGG ниже; справа – датировка образования снипа по данным компании YFull). Это – типичный снип Восточной Европы. По виду гаплотипа совершенно ясно, что он есть и у заказчика. Вопрос – сможем ли мы по виду гаплотипа предсказать некоторые нижестоящие снипы?

В базе данных (Проект FTDNA I2a имеются 245 гаплотипов в 67-маркерном формате. На дереве, построенном из всех этих гаплотипов, гаплотип ID00004 находится справа (помечен символом Х), он относится к родственной большой ветви, которая занимает большую часть правой, компактной ветви дерева. Компактная – это значит относительно молодая ветвь, по сравнению с более рыхлыми ветвями. Иначе говоря, компактная ветвь свидетельствует о ее относительно недавнем происхождении. Расчеты показывают, что этой ветви примерно 2300 лет, то есть она образовалась в конце прошлой эры. Практически все гаплотипы этой ветви сопряжены со снипом CTS10228/L147.2. Но у нее есть и нисходящие снипы, например
L147.2 > YP204/S17250 >Z16971
L147.2 > YP204/S17250 >Y4882 > A811
L147.2 > YP204/S17250 >Y4882 > A1328
L147.2 > YP204/S17250 >A356/Z16983
L147.2 > Y4460 > Y3118
L147.2 > Z17855
Которые в 67-маркерных гаплотипах на дереве не разделяются. Точнее, возможно, они могут расходиться по разным частям ветви, но слишком мало носителей гаплотипов в Проекте определяли у себя столь глубокие снипы. Их будет определять московская Лаборатория ДНК-генеалогии.

Есть еще одна причина, почему перечисленные субклады не разделяются на дереве гаплотипов – практически все они образовались почти одновременно (примерно 2100 лет назад) с родительским снипом CTS10228 (примерно 2300 лет назад). Так что их не разделяет не только 67-маркерное дерево, но и 111-маркерное дерево этой отдельной ветви:

Мы видим, что и на 111-маркерном дереве нет выраженных ветвей, которые можно было бы отнести к конкретным снипам. Базовый гаплотип ветви в 67-маркерном формате:
13 24 16 11 14 15 11 13 13 13 11 31 – 17 8 10 11 11 25 15 20 32 12 14 15 15 – 10 10 21 21 15 12 18 18 34 35 11 10 – 11 8 15 15 7 12 10 8 11 9 12 22 22 16 10 12 12 12 7 10 30 21 13 14 10 13 11 11 12 9
(выделены 8 мутаций от представленного гаплотипа). Это означает, что гаплотип удален от предкового на 8/0.12 = 67 → 72 условных поколения, то есть примерно на 1800 лет. Это, конечно, примерно, так как по двум гаплотипам точных расчетов не бывает – одна случайная мутация в представленном гаплотипе сдвинет расчетное расстояние больше, чем на 200 лет. В любом случае ясно, что предки заказчика вели ДНК-линию ID00004 практически напрямую от общего предка данной ветви.
История гаплогруппы/субклада I2a-L147.2 c возрастом примерно 2300 лет неоднократно обсуждалась на Переформате. Гаплогруппу I2a находили в захоронениях в центральной Европе с датировками 7000-5000 лет назад, затем примерно 4500 лет назад она из центральной Европы исчезла, и разошлась на две ветви. Одна (точнее, несколько ветвей, см. первую диаграмму выше) передвинулась (или бежала) на Британские острова, и общие предки этой совокупности ветвей уходят примерно на 4500-5000 лет назад. Другая ветвь субклада I2a-L147.2, прошла в Европе бутылочное горлышко популяции, возродилась около 2300 лет назад, на Дунае-Карпатах, и разошлась по всей Восточной Европе в первой половине I тыс. нашей эры. Возможно, это ее историки и археологи приняли за «происхождение славян», и это о ней идет речь в древних летописях, таких как Повесть временных лет. Если ПВЛ права, то славяне гаплогруппы I2a-L147.2 вышли из Норика, в восточных Альпах, к северу от северной части Адриатического моря. Тавриски, жители Норика, известны со второй половины I тыс. до н.э.
Если бы ID00004 сделал себе тест BigY (или другой геномный тест на Y-хромосому), то результат бы близок (или идентичен) следующему (здесь выделены вышестоящие снипы субклада I2a-CTS10228/L147.2). Остальные отмеченные снипы относятся к еще более вышестоящим гаплотипам, как, например, M42 – сводная гаплогруппа ВТ; V9, V52 и V189 – это всё сводная гаплогруппа СТ; М89 – гаплогруппа F, L15 и L16 – сводная гаплогруппа IJK, L41 – гаплогруппа I, и так далее. Как видим, единственное полезное, что дал здесь тест BigY (за 599 долларов) – это идентификация самого нижнего снипа I2a-CTS10228. Более нижестоящих снипов здесь нет, а приведенный выше снип Z17855 в списке ниже отрицательный (четвертый от конца).
Приведенный ниже набор снипов взят из открытой базы данных, он принадлежит потомку польского князя Святополк-Четвертинского, по легендам – из ополячившихся Рюриковичей. Кстати, князь Борис Антонович Четвертинский (1784-1865), участник наполеоновский войн, полковник русской армии – из того же польского рода. Большинство этих снипов, если не все, должны быть найдены и у ID00004. Впрочем, все – это только если глубокий субклад окажется точно таким же, как и у Четвертинского. Но интересно, что глубоких снипов ниже L147.2 у Четвертинского не обнаружено, самый последний, терминальный, это CTS10228, то есть L147.2.
CTS4002+, CTS4039+, CTS4088+, CTS410+, CTS4437+, CTS4848+, CTS4982+, CTS5044+, CTS5375+, CTS5650+, CTS5727+, CTS5884+, CTS5908+, CTS5966+, CTS5985+, CTS623+, CTS6265+, CTS674+, CTS6932+, CTS7175+, CTS7213+, CTS7218+, CTS7329+, CTS7831+, CTS8239+, CTS8333+, CTS8345+, CTS8420+, CTS8486+, CTS88+, CTS8876+, CTS9240+, CTS9264+, CTS9349+, CTS10058+, CTS10228+ , CTS10834+, CTS10936+, CTS11030+, CTS11441+, CTS11768+, CTS1293+, CTS176+, CTS1846+, CTS2193+, CTS2514+, CTS3296+, CTS3517+, CTS3654+, F1209+, F3145+, F3692+, F719+, L132+, L15+, L16+, L178+, L350+, L403+, L41+, L460+ , L468+, L470+, L498+, L578+, M42+, M423+ , M438+, M89+, M94+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P143+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P316+, P38+, PAGES00026+, PAGES00081+, PF2591+, PF2608+, PF2611+, PF2615+, PF2747+, PF2748+, PF2749+, PF2770+, PF3562+, PF3574+, PF3626+, PF3638+, PF3639+, PF3640+, PF3641+, PF3644+, PF3651+, PF3652+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3801+, PF3802+, PF3804+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF3966+, PF4058+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, L621+ , L68+, L748+, L751+, L755+, L756+, L758+, L772+, M168+, M170+ , M213+, M235+, M258+, M294+, M299+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, M423+, S7721+, V19-, P41-, Z17694-, Z2103-, Z2109-, M3453-, S2348-, S4442-, P61-, L584-, CTS3802-, DF41-
Поскольку речь зашла о Рюриковичах, напомним, что у группы князей Российского дворянского собрания – гаплогруппа N1c1, у Четвертинского (и у заказчика) – гаплогруппа I2a. Рюрик, если такой вобще существовал, мог иметь только одну гаплогруппу. Так что вопрос о многочисленных Рюриковичах пока остается открытым, если у всех у них есть документальные основания утверждать, что они Рюриковичи. Насколько мне известно, документальных оснований ни у кого нет, но есть устоявшиеся мнения специалистов по княжеским генеалогиям. Причем наверняка у польских генеалогов мнение одно, а у российских – мнение другое. Пусть они между собой и разбираются, но ДНК-генеалогия предоставляет им новые данные для расмотрения.
Возможно, московская Лаборатория ДНК-генеалогии сможет идентифицировать у ID00004 нижестоящие (по отношению к CTS1028) снипы, и понять, в каких регионах они преимущественно выражены. Тогда персональную генеалогию можно будет еще далее углубить.
KLIN ID00019
Представлен 111-маркерный гаплотип, сообщено, что гаплогруппа I2b, субклад I2b1, снип М223.
15 23 15 10 15 15 11 14 12 14 12 32 16 8 9 11 11 25 14 20 28 11 14 14 15 11 10 19 21 15 14 18 21 32 40 12 10 11 8 16 16 8 11 10 8 10 9 12 22 22 15 11 12 12 15 9 14 27 21 11 13 12 12 11 12 12 11 32 13 8 15 10 24 27 16 12 11 14 11 12 9 13 11 10 10 12 32 10 12 22 13 11 10 20 15 23 9 24 14 12 14 27 12 21 18 12 15 17 9 12 11
В представленных данных использована устаревшая номенклатура 2010 года, с тех пор снип М223 относят к субкладу I2a2a (см. диаграмму ниже, в сокращенном виде). В правой колонке представлены (в годах, лет назад) примерные датировки возникновения древнейших и некоторых других снипов (которые здесь представляют для нас интерес), рассчитанные компанией YFull по геномным данным.

Сиреневым цветом выше обозначен субклад I2a-L147.2, наиболее распространенный в гаплогруппе I в Восточной Европе – от Греции до Прибалтики, включая Россию, Украину, Белоруссию, страны бывшей Югославии; красной строкой выделен субклад представленного гаплотипа, I2a-M223. Сам по себе субклад древний, ему 17400 лет, и, конечно, у него имеются нижестоящие суклады, к выявлению которых мы в настоящей Интерпретации приблизимся. Желтым цветом выделены субклады, которые могут быть у ID00019, как будет показано ниже.
Попытаемся определить место представленного гаплотипа на общем дереве 111-маркерных гаплотипов гаплогруппы I2 из Проекта I FTDNA . Для этого возьмем все 63 111-маркерных гаплотипов гаплогруппы I2 Проекта, добавим к ним представленный гаплотип ID00019, и построим дерево из полученных 64 гаплотипов с помощью профессиональной компьютерной программы (PHYLIP). Цель этого эксперимента – выяснить, в окружении каких гаплотипов и из каких субкладов окажется представленный гаплотип. Дерево этих 111-маркерных гаплотипов гаплогруппы I2 приведено ниже.

Положение представленного гаплотипа обозначено на дереве индексом Х. Это – отдельная, хорошо выраженная ветвь из семи подобных по виду, предположительно родственных гаплотипов. Для сведения, базовый (предковый) гаплотип всего дерева I2 следующий
13 24 16 10 13 15 11 13 12 13 11 30 17 8 10 11 11 25 15 20 29 12 14 15 15 10 10 19 20 15 14 17 18 34 36 12 10 11 8 15 16 8 11 10 8 11 9 11 21 22 16 11 12 12 15 8 13 25 21 11 13 11 13 11 12 12 11 30 14 8 15 11 25 27 18 12 11 12 12 12 9 12 11 10 11 12 31 11 12 22 14 11 10 22 15 20 11 23 16 11 15 25 12 22 18 12 14 17 9 12 11
Представленный гаплотип отличается от него на 70 мутаций. Это эквивалентно 70/0.198 = 354 → 537 условных поколений, или примерно 13425 лет между ними (0.198 – это константа скорости мутации для 111-маркерных гаплотипов, стрелка – поправка на возвратные мутации). Иначе говоря, общий предок гаплогруппы чрезвычайно удален по времени от ID00019.
Все 64 гаплотипа имеют 3906 мутаций от базового гаплотипа, это соответствует времени, когда жил общий предок современных носителей этого гаплотипа, и которое составляет в данном случае 3906/64/0.198 = 308 → 440 условных поколений (по 25 лет каждое, под этот срок калибровались константы скорости мутаций), или 11000±1100 лет назад. Ясно, что он жил намного позже времени образования субклада I2 (примерно 27500 лет назад, определено по снипам компанией YFull), и это свидетельствует, что гаплогруппа I2 прошла около 11000 лет назад жесткое бутылочное горлышко популяции, возможно, связанной с периодом оледенения в Европе. Общий предок заказчика и гаплогруппы I2 жил примерно (13425+11000)/2 = 12200 лет назад, то есть еще до прохождения бутылочного горлышка.
Мы видим, что представленный гаплотип находится на одной ветви с гаплотипами под номерами 39, 44-46, 51, 52. Базовый гаплотип этой ветви
15 23 15 10 15 15 11 13 12 14 12 32 15 8 10 11 11 25 14 20 28 11 14 14 15 11 10 19 21 14 14 17 19 33 39 12 10 11 8 15 16 8 11 10 8 10 9 12 21 22 15 11 12 12 14 9 13 27 20 11 13 12 12 11 12 12 11 31 13 8 15 11 24 27 16 12 11 13 11 13 9 12 11 10 11 12 32 10 12 22 13 11 10 20 15 22 9 23 14 12 14 28 12 21 18 12 15 17 9 12 11
Отличается на 23 мутации (отмечены) от представленного гаплотипа, то есть гаплотип удален от предкового для данной ветви на 23/0.198 = 116 → 132 условных поколения (по 25 лет), то есть примерно на 3300 лет. В то же время все семь гаплотипов ветви имеют 163 мутации от показанного выше базового гаплотипа, то есть общий предок этой ветви жил 163/7/0.198 = 118 → 134 условных поколения, то есть 3350±425 лет назад. Мы видим, что эти две величины совпадают практически идеально (3300 и 3350 лет, тем более в пределах погрешности расчетов). Это означает, что представленный гаплотип почти идеально вписывается в данную ветвь из семи гаплотипов, и соответствует ей и по гаплотипу, и по удалению от нее, и по расчитанному возрасту общего предка ветви. Так что ветвь гаплотипа ID00019 мы нашли.
Посмотрим, к каким субкладам относятся родственные (в ДНК-генеалогическом смысле) гаплотипы, под номерами 39, 44, 45, 46, 51 и 52. Ближе всего на дереве к представленному гаплотипу сидит гаплотип 39 (Англия). У него снип Р95, с возрастом 3100 лет (выделен желтым цветом на диаграмме в начале этого материала). Это практически совпадает с возрастом рассматриваемой ветви, 3350±425 лет. Далее идут гаплотипы 51 и 52 (Шотландия и Швеция), они не типированы на снипы глубже М223, как и у ID00019. Но проект определяет их к снипам не выше Z161, L801 и CTS6433. Последние три гаплотипа на ветви, 44, 45 и 46 (Англия, Португалия и Швейцария), имеют снипы соответственно Z79, CTS6433 и CTS6433, с возрастом 2400, 3800 и 3800 лет. Таким образом, и ветвь ID00019 относительно молодая, и снипы на ней по возрасту соответствуют, учитывая, что и возраст снипов определен с определенной погрешностью.
Подводя итоги, следует заключить, что снипы ID00019 должны быть в ареале снипов, обозначенных желтым цветом на диаграмме снипов в начале Интерпретации, и скорее всего среди снипов P95, CTS6433 и Z79. Московская лаборатория ДНК-генеалогии планирует определять все эти снипы.
Исторический путь к представленному гаплотипу примерно следующий. Гаплогруппа I образовалась около 43 тысяч лет назад (из сводной гаплогруппы IJ), около 28 тысяч лет она разошлась на гаплогруппы I1 и I2, и из последней (как и из первой) образовалось множество нижестоящих субкладов. Дальнейшая история гаплогруппы I2 описана в предшествующих Интерпретациях; в итоге большинство носителей гаплогруппы I2a из Восточной Европы относятся к нисходящим ветвям субклада L147.2, общий предок которых жил в конце I тыс. до н.э., примерно 2300 лет назад. ID00029 – представитель другой, параллельной ветви, М223, но относительно недавних субкладов, общий предок которых жил во второй половине II тыс. до н.э., примерно 3300 лет назад.
Приложение . Здесь приводятся детальные геномные данные по составу снипов, обнаруженных у носителей гаплотипов с ветви ID00029. У него должны быть практически такие, за исключением, возможно, терминальных снипов. Но, возможно, и они те же, как в нескольких приведенных случаях. Выделены снипы от М223 и ниже.
Kit No. 227059 (Англия, гаплотип 39 на ветви): CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10362+, CTS109+, CTS11358+, CTS11441+, CTS11575+, CTS11726+, CTS125+, CTS12632+, CTS1977+, CTS1996+, CTS2193+, CTS2392+, CTS2514+, CTS2536+, CTS3296+, CTS3326+, CTS3331+, CTS3431+, CTS3517+, CTS3536+, CTS3654+, CTS3662+, CTS3868+, CTS3996+, CTS4039+, CTS4088+, CTS429+, CTS4314+, CTS4348+, CTS4364+, CTS4368+, CTS4443+, CTS4740+, CTS4848+, CTS4982+, CTS5286+, CTS5318+, CTS5457+, CTS5532+, CTS5650+, CTS5727+, CTS5908+, CTS6135+, CTS6136+, CTS616+, CTS6265+, CTS6331+, CTS6383+, CTS674+, CTS6800+, CTS6907+, CTS6932+, CTS7329+, CTS7331+, CTS7502+, CTS7682+, CTS7831+, CTS7865+, CTS7922+, CTS7933+, CTS7934+, CTS8243+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS8980+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, CTS9828+, F1046+, F1209+, F1302+, F1320+, F1329+, F1450+, F1460+, F1704+, F1714+, F1753+, F1767+, F2048+, F2075+, F2142+, F2155+, F2302+, F2345+, F2366+, F2402+, F2587+, F2688+, F2710+, F2794+, F2837+, F2985+, F2993+, F3111+, F3136+, F3335+, F3368+, F3402+, F3556+, F3692+, F4188+, F719+, F922+, L132+, L15+, L16+, L34+, L35+, L350+, L37+, L403+, L460+, L468+, L470+, L498+, L578+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M139+, M168+, M170+, M223+ , M235+, M294+, M42+, M89+, M94+, P123+, P124+, P126+, P127+, P130+, P135+, P136+, P138+, P14+, P141+, P145+, P146+, P148+, P151+, P158+, P159+, P160+, P166+, P187+, P214+, P215+, P216+, P217+, P219+, P220+, P221+, P222+, P223+, P305+, P95+, PAGES00026+, PAGES00081+, PF1016+, PF1029+, PF1031+, PF1040+, PF1046+, PF1061+, PF1092+, PF1097+, PF110+, PF1203+, PF1269+, PF1276+, PF192+, PF210+, PF212+, PF223+, PF234+, PF258+, PF2591+, PF2593+, PF2599+, PF2608+, PF2611+, PF2615+, PF2624+, PF263+, PF2643+, PF272+, PF2745+, PF2747+, PF2748+, PF2749+, PF2770+, PF278+, PF292+, PF316+, PF325+, PF342+, PF3515+, PF3517+, PF3518+, PF3534+, PF3560+, PF3561+, PF3562+, PF3573+, PF3574+, PF3578+, PF3586+, PF3588+, PF3590+, PF3594+, PF3595+, PF3596+, PF3600+, PF3604+, PF3605+, PF3607+, PF3611+, PF3612+, PF3616+, PF3618+, PF3621+, PF3623+, PF3625+, PF3626+, PF3634+, PF3639+, PF3640+, PF3641+, PF3642+, PF3644+, PF3651+, PF3652+, PF3654+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3686+, PF3694+, PF3780+, PF3798+, PF3800+, PF3801+, PF3802+, PF3804+, PF3806+, PF3807+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF500+, PF667+, PF719+, PF725+, PF779+, PF796+, PF803+, PF815+, PF821+, PF840+, PF844+, PF892+, PF937+, PF951+, PF954+, PF970+, V168+, V186+, V189+, V205+, V221+, V241+, V250+, V52+, V9+, YSC0000056+, YSC0000256+, YSC0000260+, YSC0000265+, YSC0000267+, YSC0000272+, YSC0000280+, YSC0000281+, YSC0000298+, YSC0000300+, Z161+ , Z162+, Z163+, Z164+, Z165+, Z168+, Z170+, Z172+, Z174+, Z175+, Z176+, Z177+, Z178+, Z179+, Z181+, Z183+, Z184+, Z186+, Z188+, Z189+, Z77+, CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10834+, CTS11441+, CTS1977+, CTS2193+, CTS2392+, CTS2514+, CTS3296+, CTS3326+, CTS3517+, CTS3654+, CTS4039+, CTS4088+, CTS429+, CTS4348+, CTS4437+, CTS4848+, CTS4982+, CTS5286+, CTS5650+, CTS5727+, CTS5884+, CTS5908+, CTS6136+, CTS616+, CTS623+, CTS6265+, CTS6331+, CTS674+, CTS6932+, CTS7329+, CTS7331+, CTS7682+, CTS7831+, CTS7865+, CTS7934+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, F1209+, F3692+, F719+, L132+, L15+, L16+, L181+, L34+, L35+, L350+, L36+, L37+, L403+, L41+, L460+, L468+, L470+, L498+, L578+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M168+, M170+, M213+, M223+, M235+, M258+, M294+, M299+, M42+, M438+, M89+, M94+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P143+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P214+, P216+, P217+, P218+, P219+, P220+, P221+, P222+, P223+, P316+, P38+, P95+ , PAGES00026+, PAGES00081+, PF2591+, PF2608+, PF2611+, PF2615+, PF2747+, PF2748+, PF2749+, PF2770+, PF3562+, PF3574+, PF3626+, PF3639+, PF3640+, PF3641+, PF3644+, PF3651+, PF3652+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3801+, PF3802+, PF3804+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, YSC0000281+, Z161+ , Z163+, Z164+, Z165+, Z168+, Z170+, Z174+, Z175+, Z176+, Z178+, Z179+, Z184+, Z186+, Z188+, P37-, S2348-, S4442-
Kit No. 173448 (Англия, гаплотип 44): Z77+, L800+, L801+ , V218+, Z186+, Z78+ , Z79+, L1198+ , Z190+, Z185+, Z171+, Z166+, CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10834+, CTS11441+, CTS2134+, CTS2193+, CTS2392+, CTS2514+, CTS3296+, CTS3326+, CTS3517+, CTS3654+, CTS4039+, CTS4088+, CTS429+, CTS4348+, CTS4437+, CTS4848+, CTS4982+, CTS5286+, CTS5650+, CTS5727+, CTS5884+, CTS5908+, CTS6136+, CTS616+, CTS623+, CTS6265+, CTS6331+, CTS6433+ , CTS674+, CTS6932+, CTS7329+, CTS7331+, CTS7682+, CTS7831+, CTS7865+, CTS7934+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, F1209+, F3692+, F719+, L104+, L132+, L15+, L16+, L181+, L34+, L35+, L350+, L36+, L37+, L403+, L41+, L460+, L468+, L498+, L578+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M168+, M170+, M213+, M223+ , M235+, M258+, M294+, M299+, M42+, M438+, M89+, M94+, P108+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P143+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P214+, P216+, P217+, P218+, P219+, P220+, P221+, P222+, P223+, P305+, P316+, PAGES00026+, PAGES00081+, PF2591+, PF2608+, PF2611+, PF2615+, PF2624+, PF2747+, PF2748+, PF2749+, PF2770+, PF3562+, PF3574+, PF3626+, PF3639+, PF3640+, PF3641+, PF3644+, PF3651+, PF3652+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3801+, PF3802+, PF3804+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, PK1+, SRY10831+, V221+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, Z163+, Z164+, Z165+, Z168+, Z170+, Z174+, Z175+, Z178+, Z179+, Z184+, Z185+ , Z188+, Z190+, Z79+ , L707+, M223+, M253-, M26-, M284-, M379-, P78-, P95- , S2348-, S4442-
Kit No. 18187 (Португалия, гаплотип 45): CTS674+, CTS6800+, CTS6907+, CTS6932+, CTS7329+, CTS7331+, CTS7502+, CTS7682+, CTS7831+, CTS7865+, CTS7922+, CTS7933+, CTS7934+, CTS8243+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS8980+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, CTS9828+, F1046+, F1209+, F1302+, F1320+, F1329+, F1450+, F1460+, F1704+, F1714+, F1753+, F1767+, F2048+, F2075+, F2142+, F2155+, F2302+, F2345+, F2366+, F2402+, F2587+, F2688+, F2710+, F2794+, F2837+, F2985+, F2993+, F3111+, F3136+, F3335+, F3368+, F3402+, F3556+, F3692+, F4188+, F719+, F922+, L132+, L15+, L16+, L34+, L35+, L350+, L37+, L403+, L41+, L460+, L468+, L470+, L498+, L578+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M139+, M168+, M170+, M223+ , M235+, M294+, M42+, M89+, M94+, P123+, P124+, P126+, P127+, P130+, P135+, P136+, P138+, P14+, P141+, P145+, P146+, P148+, P151+, P158+, P159+, P160+, P166+, P187+, P214+, P215+, P216+, P217+, P219+, P220+, P221+, P222+, P223+, PF1016+, PF1029+, PF1031+, PF1040+, PF1046+, PF1061+, PF1092+, PF1097+, PF110+, PF1203+, PF1269+, PF1276+, PF192+, PF210+, PF212+, PF223+, PF234+, PF258+, PF2591+, PF2593+, PF2599+, PF2608+, PF2611+, PF2615+, PF2624+, PF263+, PF2643+, PF272+, PF2745+, PF2747+, PF2748+, PF2749+, PF2770+, PF278+, PF292+, PF316+, PF325+, PF342+, PF3515+, PF3517+, PF3518+, PF3534+, PF3560+, PF3561+, PF3562+, PF3573+, PF3574+, PF3578+, PF3586+, PF3588+, PF3590+, PF3594+, PF3595+, PF3596+, PF3600+, PF3604+, PF3605+, PF3607+, PF3611+, PF3612+, PF3616+, PF3618+, PF3621+, PF3623+, PF3625+, PF3626+, PF3634+, PF3639+, PF3640+, PF3641+, PF3642+, PF3644+, PF3651+, PF3652+, PF3654+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3686+, PF3694+, PF3780+, PF3798+, PF3800+, PF3801+, PF3802+, PF3804+, PF3806+, PF3807+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, PF500+, PF667+, PF719+, PF725+, PF779+, PF796+, PF803+, PF815+, PF821+, PF840+, PF844+, PF892+, PF937+, PF951+, PF954+, PF970+, V186+, V189+, V205+, V52+, V9+, YSC0000056+, YSC0000256+, YSC0000260+, YSC0000265+, YSC0000267+, YSC0000272+, YSC0000280+, YSC0000281+, YSC0000298+, YSC0000300+, Z161+ , Z162+, Z163+, Z164+, Z165+, M170+, M223+, M258+, P38+, L801+, Z186+, Z76+, L1316+, CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10362+, CTS109+, CTS11358+, CTS11441+, CTS11575+, CTS11726+, CTS125+, CTS12632+, CTS1996+, CTS2193+, CTS2392+, CTS2514+, CTS2536+, CTS3296+, CTS3326+, CTS3331+, CTS3431+, CTS3517+, CTS3536+, CTS3654+, CTS3662+, CTS3868+, CTS3996+, CTS4039+, CTS4088+, CTS429+, CTS4314+, CTS4348+, CTS4364+, CTS4368+, CTS4443+, CTS4740+, CTS4848+, CTS4982+, CTS5286+, CTS5318+, CTS5457+, CTS5532+, CTS5650+, CTS5727+, CTS5908+, CTS6135+, CTS6136+, CTS616+, CTS6265+, CTS6331+, CTS6383+, CTS6433+ , F3692+, F719+, L104+, L132+, L15+, L16+, L181+, L34+, L35+, L350+, L36+, L37+, L403+, L41+, L460+, L468+, L498+, L59+, L68+, L748+, L751+, L755+, L756+, L758+, L772+, L800+, M168+, M170+, M213+, M223+, M235+, M258+, M294+, M299+, M42+, M438+, M89+, M94+, P123+, P124+, P125+, P126+, P127+, P129+, P130+, P133+, P134+, P135+, P136+, P138+, P139+, P14+, P140+, P141+, P145+, P146+, P148+, P149+, P151+, P157+, P158+, P159+, P160+, P161+, P163+, P166+, P187+, P212+, P214+, P216+, P217+, P218+, P219+, P220+, P221+, P222+, P223+, P316+, PAGES00026+, PAGES00081+, PF2591+, PF2608+, PF2611+, PF2615+, PF2747+, PF2748+, PF2749+, PF2770+, L1409+, PF3833+, Z2198+, CTS8444+, CTS6497+, CTS10057+, CTS10058+, CTS10100+, CTS10125+, CTS10834+, CTS11441+, CTS2134+, CTS2193+, CTS2392+, CTS2514+, CTS3296+, CTS3326+, CTS3517+, CTS3654+, CTS4039+, CTS4088+, CTS429+, CTS4348+, CTS4437+, CTS4848+, CTS4982+, CTS5286+, CTS5650+, CTS5727+, CTS5884+, CTS5908+, CTS6136+, CTS616+, CTS623+, CTS6265+, CTS6331+, CTS674+, CTS6932+, CTS7329+, CTS7331+, CTS7682+, CTS7831+, CTS7934+, CTS8333+, CTS8345+, CTS8420+, CTS8449+, CTS88+, CTS8876+, CTS8901+, CTS9183+, CTS9240+, CTS9264+, CTS9266+, CTS9482+, CTS9782+, PF3562+, PF3574+, PF3626+, PF3639+, PF3640+, PF3641+, PF3644+, PF3651+, PF3652+, PF3657+, PF3658+, PF3660+, PF3664+, PF3666+, PF3667+, PF3669+, PF3671+, PF3672+, PF3675+, PF3677+, PF3780+, PF3800+, PF3801+, PF3802+, PF3804+, PF3809+, PF3811+, PF3812+, PF3814+, PF3815+, PF3819+, PF3820+, PF3822+, PF3836+, PF3837+, PF3849+, PF3876+, Z172+, Z174+, Z175+, Z176+, Z177+, Z178+, Z179+, P19+, Z181+, Z183+, Z184+, Z186+, V221+, V241+, V250+, YSC0000207+, YSC0000227+, YSC0000256+, YSC0000272+, Z163+, Z164+, Z165+, Z168+, Z170+, Z174+, Z175+, Z178+, Z184+, PF6464+, PF6469+, PF6470+, PF6477+, PF6479+, PF6520+, Z168+, Z170+, Z188+, Z77+, Z188+, F1209+, F3406-, M161-, Z190-, Z63-, Z79-, L1317-, Z171-, S2348-, S4442-, Z185-, Z187-, L1201-, P30-, P37-, Z180-, PF4225-, DF5-, PF3292-, CTS661-, L1198-, Z190- , Z2059-, Z2084-, Z2074-, L1272-, ZS6-, ZS7-, ZS11-, ZS14-, ZS15-, ZS16-, ZS18-, ZS26-, L1290-, Z78-, Z79- , M284-, M379-, P78-, P95-, L380-, M26-, M307-, M72-, M227-, M253-, M21-, Z166-
Kit No. B3411 (Швейцария, гаплотип 46): L801+, CTS6433+, M223+ , L1201-, Z190-, Z78-, CTS5332-
Kit No. 23157 (Шотландия, гаплотип 51): P19+, M170+, M258+, M223+ , P19+, P38+, M284-, M379-, P78-, P95-, P30-, P37-, M227-, M253-, M26-, M307-, M72-, M21-, M161-
Kit No. 174472 (Швеция, гаплотип 52): M284-, L126-, M379-, P78-, P95-
Гаплогруппа I2c
Эту гаплогруппу имел один человек, с 67-маркерным гаплотипом.
KLIN ID00005
Сообщено, что субклад I2c-L596, глубже субклад в FTDNA не определяли.
14 24 15 10 12 13 11 13 12 12 11 28 – 18 8 8 11 11 24 15 21 31 11 11 15 15 – 10 10 19 21 15 13 17 17 34 34 14 10 12 8 16 16 8 13 10 8 10 9 12 21 21 16 11 12 13 13 8 14 25 21 12 13 11 13 11 12 12 11
Поскольку глубже, чем I2c-L596, субклад не определяли, то цепочка фактически определенных субкладов в гаплогруппе I довольно короткая: I-M170 > I2-M438 > I2c-L596. Определение в FTDNA проведено правильно, как показывает позиция представленного гаплотипа на дереве 67-маркерных гаплотипов. Но возможны следущие нисходящие субклады, которые пока не определяли:

Ниже приведено дерево 67-маркерных гаплотипов гаплогруппы I-M170 (гаплотипы взяты из Проекта FTDNA). Всю левую часть дерева занимают гаплотипы гаплогруппы I1-M253, c общим предком 3690±370 лет назад (хотя эта гаплогруппа возникла примерно 27500 лет назад, но она прошла бутылочное горлышко популяции примерно 3500-4000 лет назад, как и все субклады гаплогруппы I в этот период времени, в целом между 4000 и 5000 лет назад), они имеют вид плотной, компактной ветви. Гаплогруппа I2 занимает правую часть дерева, там расположены гаплотипы субкладов I2a, I2b, и I2c, последние – в правой верхней части дерева, и отделены от остальных высокой узкой ветвью, которя к I2c не относится. На представленный гаплотип ID00005 указывает угол квадратика с буквой Х. Эта ветвь, одна из трех ветвей субклада I2c-L596, приведена ниже в увеличенном виде, и представленный гаплотип опять помечен крестиком.
Теперь о ветви из 9 гаплотипов (три из Грузии [под номерами 427, 451 и 452], два из Белоруссии , по одному из Литвы , Австрии и Турции ). Базовый гаплотип этой ветви (мутационные отличия от представленного гаплотипа отмечены)
14 24 15 10 12 13 11 13 11 13 11 29 – 18 8 8 11 11 24 15 20 32 11 14 15 16 – 11 10 19 21 15 13 17 19 34 36 12 10 12 8 16 16 8 12 10 8 10 9 12 21 21 16 11 12 13 14 8 13 25 20 12 13 11 13 11 12 12 11
Все 9 гаплотипов ветви имеют 123 мутации от этого базового гаплотипа, что означает, что общий предок ветви жил 123/9/0.12 = 114 → 129 условных поколений назад, или 3225±430 лет назад. Представленный гаплотип отличается от базового на 17 мутаций (ряд мутаций считаются по особым правилам), и это дает примерно то же самое время от общего предка, в пределах погрешности расчета. Это показывает, что ID00005 – прямой потомок общего предка ветви гаплогруппы I2c, который жил примерно в конце II тыс. до н.э.

Ниже показаны три основные ветви субклада I2c-L596, которые соответствуют семи его нижестоящим субкладам (см. диаграмму выше). Представленный гаплотип определенно входит в один из них, но в какой – мы пока не знаем. У каждого – своя история, которая пока нам тоже неизвестна (та, что известна, приведена ниже). Как мы видим, география ветви ID00005 весьма обширна – от Австрии, Беларуси и Литвы до Турции и Грузии, но более тонкое подразделение ветвей по нижестоящим субкладам в итоге позволит ее уточнить. Проекты FTDNA в этом отношении пока не помощники, в них этих более тонких субкладов нет, но московская Лаборатория ДНК-генеалогии будет их определять.
Чтобы понять, насколько сложной и трагической была история субклада L596 и его нижестоящих субкладов, заметим, что сам субклад L596 возник примерно 21300 лет назад (как показывает лесенка его снипов в Y-хромосоме), но он в ходе истории Европы то почти полностью погибал, то снова возрождался из немногих выживших носителей L596. В итоге предок подветви ID00005 жил примерно 3225 лет назад, соседней подветви (из семи гаплотипов) на диаграмме выше – примерно 2475 лет назад, верхней подветви (из девяти гаплотипов) – 2800 лет назад, и всех трех ветвей вместе – 5950±645 лет назад. А геномный анализ показывает, что нижестоящий субклад I2c1 был образован 15500 лет назад; следующий ниже субклад, I2c1a-L1251 – 10100 лет назад, про остальные данных пока нет. Мы видим, что пока обнаружены три ветви L596, возраст каждой – около 3200-2500 лет назад.

Как уже говорилось, показанная ветвь по географии смешанная, но с уклоном в Восточную Европу и Кавказ (Австрия, Беларусь, Литва, Грузия, Турция). Вторая ветвь – западная (Англия, Франция, Италия, Нидерланды). Третья – тоже западная (Англия, Шотландия, Ирландия, Дания, Германия).
Итак, подводим итоги по состоянию наших знаний на сегодняшний день. Гаплогруппа I – очень древняя, образовалась более 40 тысяч лет назад, где – пока неизвестно, но скорее всего в Европе. Около 30 тысяч лет назад она разошлась на субклады I1 и I2, и последняя разошлась на субклады I2a, I2b и I2c-L596 примерно 21300 лет назад. Гаплогруппы I и I2 обнаружены в древних ископаемых костных останках в Центральной Европе и в Швеции с датировками 7 тысяч лет назад. Немногим позже жил общий предок трех ветвей I2c-L596, потомки которого живут в настоящее время от Британских островов до Турции. Но примерно 4500 лет назад жизнь носителей гаплогруппы I, как и всех остальных в Европе резко нарушается, и почти все гаплогруппы (кроме R1b) из Европы исчезают, или откатываются на периферийные районы Европы, часть уходит в Переднюю Азию и на Кавказ.
Эти гаплогруппы в виде отдельных представителей проходят долгий и болезненный период выживания, и выжившие начинают приумножаться, формируя общих предков новых ветвей. В их числе были и носители субклада I2c-L596, пошедшие «в рост» 3200-2500 лет назад, то есть в конце II – середине I тыс. до н.э. Почти тогда же стала возрождаться гаплогруппа I2a, общий предок которой жил 2300 лет назад в Восточной Европе, и около 5000 лет назад на Британских островах. Немногим раньше стала возрождаться гаплогруппа I1, с выжившим общим предком примерно 3700 лет назад. В этом отношении субклад I2c-L596 имеет наиболее древние корни в отношении сегодняшних носителей этой гаплогруппы – около 6000 лет назад.
Отрадно, что нижестоящие субклады L596 разделяются на соответствующие ветви гаплотипов (см. диаграмму выше). Это значит, что при выявлении этих нижестоящих субкладов в московской Лаборатории ДНК-генеалогии у достаточного количества человек (например, у полусотни) мы сможем провести более детальное отнесение гаплотипов к субкладам, и более детально разобраться с преимущественной географией субкладов.
Проба персональной интерпретации мтДНК
Безусловно, будет полезно расширить круг персональных интерпретаций на женские гаплогруппы, и для их тестирования можно анализировать мДНК как у женщин, так и мужчин. Дело в том, что мужчина получает мтДНК с кровью от матери, но дальше мтДНК уже не передает, потому что митохондрии сперматозоидов после оплодотворения (или в ходе него) разрушаются. Сперматозоид содержит около десятка митохондрий, поскольку на вращение хвостиком нужна энергия, а ее поставляют именно митохондрии. Когда сперматозоид достиг цели, то митохондрии отбрасываются. Узнаете мужчин?
В очень редких случаях митохондрия все-таки проскакивает, но это по отношению к организму будущей матери чужая митохондрия, и добром это, как правило, не кончается. А кончается нередко острыми патологиями.
Короче, мтДНК передается от матери дочерям по всей цепочке поколений, десятками, сотнями тысяч лет, миллионами лет, и так далее. А сыновьям – только на одно поколение. Сыновья передают своим сыновьям Y-хромосому, не передают никому мтДНК – ни сыновьям, ни дочерям, но передают всем свои остальные 22 хромосомы, рекомбинантные. Поэтому, повторяю, тесты на мтДНК можно проводить по ДНК как женщин, так и мужчин. В обоих случаях результат один – выявление мтДНК матери и всей предыдущей цепочки мтДНК со всеми накопленными мутациями. То, что это дает – описано во множестве изданий, и, в частности, в наиболее адаптированном для нас виде – в книгах «Интернет. Заметки научного сотрудника» (2010), на стр. 368-379, и в несколько дополненном виде – в книге «Происхождение славян» (2013), стр. 265-284.
Прежде чем начать акцию по массовой персональной интерпретации мтДНК, предлагаем пробный вариант, выполненный одним из коллег. Поскольку вариант тестовый, имя автора интерпретации пока скрыто. На данном этапе важно, чтобы продукт был признан как минимум приемлемым, а лучше – хорошим или даже отличным. Так что это – на рассмотрение потенциальных заказчиков, а также всех читателей.
Итак, в качестве пробного варианта рассмотрим следующий:

Представленные данные позволяют проследить историю рода на глубине от палеолита (каменного века) лишь до горизонта нескольких тысячелетий до н.э. Это обусловлено тем, что, во-первых, женские митохондриальные ДНК имеют значительно меньшую длину высоковариативных участков, и, следовательно, намного менее информативны. А во-вторых, представленные данные не содержат доступную детализацию – например, субклад Н1с, к которому, как будет показано ниже, относится представленная картина мутаций, содержит более 20 нижестоящих субкладов. Поэтому история рода может быть реконструирована лишь с большей степенью приблизительности, по сравнению с интерпретацией данных по Y-хроосоме. Анализ различий HVR1 и HVR2 с RSRS показывает, что представленный гаплотип относится к митохондриальной гаплогруппе H1с. В силу упомянутых выше причин предоставленный образец совпадает с данными многих других мтДНК: в базе данных familytreedna.com присутствуют носители гаплотипов с идентичными RSRS-значениями по HVR1 и HVR2, причем подавляющее их большинство относится к северной и восточной Европе, например, мтДНК для Anela Sarocka (Литва).
История гаплотипа . Женская гаплогруппа H, предположительно, возникла в Западной Азии около 30 тысяч лет назад, прибыла в Европу около 20-25 тысяч лет назад и распространилась на юго-запад континента во франко-кантабрийский регион, что, скорее всего, соответствует культуре мадлен. В период последнего ледникового максимума 20-13 тысяч лет назад большинство палеолитических поселений Северной и Центральной Европы вымерло, в связи с чем представители гаплогруппы Н в большей степени выжили лишь на севере Испании (поэтому в настоящее время данная гаплогруппа с высокой частотой, более 50%, встречается среди басков, которые сложились в данном регионе при ассимиляции синокавказскими по языку пришельцами местного автохтонного населения). Данная (ископаемая) гаплогруппа была выявлена в пещерах La Pasiega и La Chora (мадлен).
На более поздних горизонтах следует отметить раннее присутствие гаплогруппы в Северной Европе и на Русской равнине, начиная с мезо-неолита – в Сертее (Смоленская область), где в слое Сертея VIII был обнаружен носитель гаплотипа H в сочетании с мужским гаплотипом R1a1 (IV тыс. до н.э.). Данная археологическая культура может рассматриваться как дальняя периферия культуры воронковидных кубков. Также подобные гаплотипы были найдены в захоронениях V-IV тысячелетий до н.э. в донецком регионе и в могильнике Южный Олений Остров (где также была выделена и мужская гаплогруппа R1a1*).
Субклад H1c . Показатель T477C позволяет определить принадлежность гаплотипа к субкладу H1c. Предполагается, что распространение подклассов H1, H3, а также сестринской гаплогруппы V связано с внутриевропейской экспансией во франко-кантабрийском регионе после последнего ледникового максимума около 13 тысяч лет назад. Гаплогруппа H1 составляет значительную долю западноевропейских митохондриальных ДНК, также в большой степени свойственна баскам Испании (28%), португальцам (26%), жителям Андалусии (24%) и этнографической группе пасьего в Кантабрии (24%). В Северной Африке необычно высокая доля гаплогруппы H1 среди туарегов Ливии (61%), что также указывает на смежный с Испанией регион. Также мтДНК гаплогруппа H1 распространена среди других жителей Иберийского полуострова, Северной Африки и Сардинии. При этом ее носители составляют свыше 10% популяции населения во Франции, на Британских островах, в Альпах, во многих регионах Восточной Европе, и не менее 5% в прочих местах Европы.
Возраст гаплогруппы H1c около 9400 лет. И в связи с этим встает вопрос о времени и дате его перемещения в Северную и Восточную Европу. Данный субклад обнаружен у земледельцев культуры воронковидных кубков Скандинавии, 3500-2500 до н.э. (Gokhem2). В предшествующих культурах Скандинавии и Севера Европы подобного гаплотипа не находили. Учитывая появление этого субклада на севере и востоке Европы уже в мезо- неолите, в широкой полосе от Оленьего Острова и до юга Украины позволяет ставить вопрос о миграции с юга. Поскольку франко-испанский регион может рассматриваться как один из центров распространения данной гаплогруппы в предшествующую эпоху, то в качестве логичной гипотезы о появлении этого гаплотипа можно считать версию о проникновении в Северную и Восточную Европу морским путем со Средиземноморского бассейна носителей культуры кардиальной керамики (гипотеза Д.Л. Гаскевича о северопонтийском импрессо).
Она позволяет связать появление носителей H1 за пределами Западной Европы с миграциями (либо иными культурно-археологическими связями) комплекса кардиальной керамики, чьи представители могли передвигаться вдоль побережья на кожаных лодках. Гаскевич даже привел создателей мегалитических культур и кардиальной керамики импрессо Западной Европы и Средиземноморья, в среде которых встречается данный субклад, через существовавший до VI тысячелетия до н.э. Босфорский перешеек в Крым и на юг Украины через буго-днестровскую культуру.

В качестве итога анализа можно констатировать, что представленный гаплотип соответствует найденному в Gokhem2 субкладу представительницы культуры воронковидных кубков, которую многие исследователи считают индоевропейской. Материнская гаплогруппа H была найдена в районе древнего присутствия мужских гаплотипов R1a1 (Южный Олений Остров, Сертея), что позволяет считать эту гаплогруппу исконной для Русской равнины и своеобразным «спутником» мужской R1a1. Более давние связи уводят корни H1c в Средиземноморье.
Для детального исследования родословной заказчика рекомендуется сделать более детальное типирование, поскольку, напоминаем, гаплогруппа H1c содержит свыше 20 различных субкладов. Такое типирование будет вскоре проводиться московской Лабораторией ДНК-генеалогии.
В печати
Рассмотрим сначала гаплогруппу I2a1 (мутация P37.2). Для славян (и притом почти исключительно для славян!) характерна ее ветвь I2a1b (мутации L178/S32 и M423), а точнее - подветвь I2a1b3 (мутация L621/S392) и ее дочерняя I2a1b3a (L147.2). Еще точнее сказать пока сложно, потому что на наличие мутаций L621/S392 и L147.2 протестировано очень небольшое число индивидов. Помимо славян, среди них есть единичные представители Германии, Италии, Великобритании. Не исключено, что среди 58 миллионов нукледотидов в Y-хромосоме будет обнаружена еще более поздняя мутация, присущая исключительно славянской подветви.
Распространение славянской подветви гаплогруппы I2a1 лучше прослеживается не по бинарным маркерам, а по характерным для нее гаплотипам. В подветви I2a1b они высоко специфичны, позволяя с высокой надежностью выявить ее представителей. В базе данных компании FamilyTreeDNA (FTDNA), осуществляющей коммерческое тестирование для интересующихся ДНК-генеалогией, представлены все выявленные в ходе такого тестирования гаплотипы. Интересующая нас подветвь там определена как «динарский кластер» . Он распадается на две ветви - южную, собственно динарскую, и северную, представленную во всех славянских популяциях (ее правильнее было бы именовать «карпатской»).
Частоту обеих ветвех в разных популяциях по этой базе определить невозможно, поскольку представленные в ней выборки сформированы по случайному принципу (совпадение желания и возможности заказать тест ДНК), очень разнятся по размерам и непредставительны для популяций в целом.
Методически корректные выборки можно найти в международной референтной базе данных Y-гаплотипов (YHRD), созданной и поддерживаемой в криминалистических целях, а также в аналогичной по назначению референтной базе данных ДНК-маркеров населения Республики Беларусь, которую создал Центр судебных экспертиз и криминалистики Министерства юстиции Республики Беларусь[Референтная база данных частот встречаемости гаплотипов Y-STR локусов в Беларуси: http://dnkexpertiza.org/index1.html ]. Гаплотипы по некоторым выборкам, не включенным в YHRD, можно найти в научных публикациях. Все эти выборки представлены сравнительно малым, в сравнении с FTDNA, числом локусов: в коммерческих тестах используются наборы в 37, 67 и даже 111 локусов, а в криминалистических - в лучшем случае 17-локусный набор, известный как стандарт Y-Filer. Но даже этот набор позволяет уверенно распознатьгаплотипы, характерные для динарского кластера. Им присущи сравнительно небольшие генетические расстояния от модального для этого кластера гаплотипа, который в формате Y-Filer приведен в таблице 1.
Таблица 1. Модальный гаплотип динарского кластера гаплогруппы I2a1
|
Число повторов |
Гаплотипы, суммарно отличающиеся от этого профиля не более чем на 9-10 шагов (мутаций в числе повторов), в подавляющем большинстве случаев принадлежат к интересующей нас ветви I2a1. Возможная ошибка в их определении не превышает 1-2 %, что подтверждается в тех (увы, немногочисленных) случаях, когда выборки одновременно тестировались и по бинарным маркерам.
Южная ветвь динарского кластера по 67 и тем более 111 локусам выделяется неплохо, но в 17-локусном наборе Y-Filerее отличительной особенностью является лишь пониженное число повторов в локусе DYS-448: 19 (изредка даже 18) вместо 20. Разумеется, различение двух субкластеров всего по одному локусу возможно только в первом приближении, но общая картина ясна даже при такой грубой оценке. Частоты гаплотипов, предположительно принадлежащих к данному кластеру, по всем опубликованным выборкам формата Y-Filerпредставлены в таблице 2.
Таблица 2. Частота гаплотипов динарского кластера
|
Выявлено |
Субкластер с DYS448<=19 |
Субкластер с DYS448>=20 |
|||||||
|
Популяция |
гаплотипов |
Частота (%) от: |
Частота (%) от: |
||||||
|
кластера |
кластера |
||||||||
|
Южная Хорватия 1 |
|||||||||
|
Южная Хорватия 2 |
|||||||||
|
Восточная Хорватия 1 |
|||||||||
|
Сербия (сербы) 3 |
|||||||||
|
Западная Хорватия 1 |
|||||||||
|
Центральная Хорватия 1 |
|||||||||
|
Черногория (сербы) 3 |
|||||||||
|
Северная Хорватия 1 |
|||||||||
|
Румыния (4 выборки) 1 |
|||||||||
|
Македония 1 |
|||||||||
|
Сербия (закарпат. русины) 1 |
|||||||||
|
Сербия (словаки) 1 |
|||||||||
|
Западная Украина (Львов) 1 |
|||||||||
|
Беларусь (центр, юг, восток) 4 |
|||||||||
|
Беларусь (запад, север) 4 |
|||||||||
|
Северная Греция 1 |
|||||||||
|
Албания 1 |
|||||||||
|
Польша (Вроцлав) 1 |
|||||||||
|
Польша (Варшава) 1 |
|||||||||
|
Южная Польша 1 |
|||||||||
|
Россия (13 выборок) 1 |
|||||||||
|
Австрия (3 выборки) 1 |
|||||||||
|
Германия (4 выборки) 1 |
|||||||||
|
Турция (5 выборок) 1 |
|||||||||
|
Италия (11 выборок) 1 |
|||||||||
|
Источники: 1 База данных YHRD 2 Ljubković etal., 2008 3 Mirabaletal., 2010 4 База данных Y-STR локусов в Беларуси |
|||||||||
Из таблицы видно, что динарский кластер гаплогруппы I2a1 имеет максимум в балкано-карпатском регионе, а среди западных и восточных славян - в ареале лука-райковецкой культуры. И в Польше, и в России его частота падает, а в Литве и Германии он представлен на уровне, который вполне может быть объяснен славянской примесью. Это значит, что и у бастарнов, и у венетов, принесших праславянский язык из лужицко-поморского ареала, этот кластер еще отсутствовал - равно как и у древних балтов до начала их интенсивных контактов со славянами. В генофонд современных славян он попал в результате ассимиляции значительной группы автохтонного населения. Из всех неславянских народов гаплогруппа I2a1 имеет высокую частоту только у румын, представляющих собой результат смешения ранних славян (носителей пражской и ипотешти-чурелской культур) с валахами - потомками фракийского населения, роменизированного в эпоху Римской империи. При этом частота у румын и в ареале формирования славянского этноса (Львовская область, юг Беларуси) примерно одинакова. Следовательно, у валахов она изначально была не ниже, чем у ранних славян.
Показательно распространение южного субкластера. Для славянских популяций он в целом нехарактерен. Даже те немногие гаплотипы с DYS448=19, что имеются в восточнославянских и польских выборках, по остальным локусам довольно далеки от модальных значений и, вероятно, отражают самостоятельные мутации. В то же время южнославянские гаплотипы часто имеют профиль, очень близкий к модальному. Большинство их носителей явно являются потомками одного индивида, имевшего 19 повторов в локусе DYS448. Этот индивид жил позднее, чем родоначальник всего динарского кластера, но ненамного - в целом разброс гаплотипов в южном субкластере довольно велик, генетические дистанции в 6-7 шагов от предкового гапллотипа в нем нередки.
Похоже, и предковый кластер с 20 повторами, и дочерний субкластер с 19 повторами, к концу бронзового - началу железного века были распространены в основном к югу от Карпат, притом дочерний возник в южной части ареала и не успел распространиться на всю Карпатскую котловину до появления славян. После эвакуации Дакии в 271 г. н. э. он был присущ, видимо, только подданным Византии, которые практически не пересекали Дунай. Тем временем более старый и обширный субкластер с DYS448=20 проник к северу от Карпат, где и влился в состав формирующегося славянства.
Когда и где предки славян включили в свой состав группу, имевшую высокую долю «динарских» гаплотипов? Это не могло быть ранее IIв. до н.э. - времени сложения зарубинецкой культуры. Но это не могло быть и позже рубежа VII- VIIIвв., когда в процессе возвратной миграциина север сложилась лука-райковецкая культура. Иначе никак не объяснить современную частоту I2a1 у русских и поляков, сформировавшихся на тех территориях, где эта гаплогруппа изначально отсутствовала.
Здесь возможны два сценария. Носителями I2a1 могли быть представители одной из местных культур, принявших участие в сложении зарубинецкой культуры или одной из позднезарубинецких групп. Речь может идти о милоградцах-неврах, борисфенитах-скифах или юхновцах-меланхленах. При втором сценарии этот компонент генофонда был приобретен уже в процессе расселения формирующихся славян, при ассимиляции ими остатков черняховской культуры на Украине или романизированных фракийцев в Карпатской котловине и на среднем Дунае.
Наличие милоградского субстрата в зарубинецкой культуре (особенно в ее верхнеднепровском варианте) вполне вероятно, хотя оценки его роли у разных исследователей расходятся. Позднескифский субстрат явственно ощутим в среднеднепровском варианте. Значительный юхновский компонент принял участие в сложении позднезарубинецких групп - почепской, картамышевской и типа Грини - Абидня, при том, что последний сыграл решающую роль в сложении киевской культуры. Но этот сценарий плохо объясняет высокое сходство гаплотипов северного и южного субкластеров. То, что известно о происхождении юхновской культуры, не позволяет говорить о наличии в ней компонента, общего с культурами балкано-карпатского региона. В милоградской и лесостепной скифской культурах таким компонентом мог быть отчетливый юго-западный импульс, который фиксируется при формировании чернолесской культуры в начале Iтысячелетия до н. э. Население, родственное протофракийским создателям штампованной и каннелированной керамики, вполне могло принести «динарские» гаплотипы в украинские лесостепи и затем по эстафете преемственности передать их скифам и милоградцам. Но носители этих гаплотипов на протяжении нескольких столетий должны были существовать в изоляции от основного фракийского мира, накопив заметное число специфических мутаций. Между тем на территории Беларуси и России нет никакой особой специфики в I2a1-гаплотипах.
При втором, более вероятном сценарии донорами северодинарских гаплотипов служат культура карпатских курганов, поглощеная пражской в процессе экспансии (она отождествляется с племенем карпов), и остаточные группы дако-романского населения в Карпатской котловине. Когда славяне смешались с ними в пределах Аварского каганата, частота северного субклада закрепилась у них на уровне около 20%. Затем они частично унесли свои гаплотипы в лесную зону, где по мере смешения с местными популяциями частота снизилась. Те же, кто ушел дальше на юг, в пределы Византийской империи, столкнулись там с автохтонами, имевшими высокую концентрацию южнодинарских гаплотипов. Именно этому субстрату хорваты, сербы и отчасти македонцы обязаны высокой частотой I2a1 в целом, значительно превышающей общеславянский уровень. Славяне, осевшие южнее Дуная, в возвратной миграции почти не участвовали, потому и южнодинарских гаплотипов в лесной зоне оказалось немного. Единственными, кто массово принес эти гаплотипы на север от Дуная, были славянские предки австрийцев, а также валахи - предки румын.